Zwei der aufregendsten und vielseitigsten genetischen Werkzeuge, die in den letzten 30 Jahren entwickelt wurden, sind die Cre-lox- und FLP-FRT-Technologien. Beide erlauben es, den Ort und den Zeitpunkt der Genexpression genau zu regulieren. In diesem Artikel wird kurz beschrieben, wie die beiden Systeme funktionieren und in Mausmodellen verwendet werden können.
Das Cre-lox-System
Die Cre-lox-Technologie wurde in den 1980er Jahren eingeführt (Sauer und Henderson 1988; Sternberg und Hamilton 1981) und von DuPont Pharmaceuticals patentiert. Es wurde erfolgreich in Hefen, Pflanzen, Säugetierzellkulturen und Mäusen angewendet (Araki et al. 1987). Es basiert auf der Fähigkeit des P1-Bakteriophagen-Cyclisierungsrekombinations- (Cre) -Rekombinase-Gens (cre), eine Rekombination zwischen Paaren von loxP-Stellen zu bewirken. Eine solche Rekombination in einer „Cre-lox“ -Maus (siehe unten) kann ein interessierendes Gen entweder aktivieren oder inaktivieren.
Um die Cre-Lox-Technologie zu verwenden, muss ein Forscher eine Cre-Lox-Maus herstellen, typischerweise durch Züchten einer Cre-Maus zu einer loxP-Maus. Eine Cre-Maus enthält ein Cre-Rekombinase-Transgen unter der Leitung eines gewebespezifischen Promotors; Eine loxP-Maus enthält zwei loxP-Stellen, die ein interessierendes genomisches Segment, den „Floxed“ -Locus, flankieren. Typischerweise werden Cre- und loxP-Mäuse unter Verwendung transgener Technologie hergestellt (Nagy 2000). Abhängig von den Promotoren und anderen regulatorischen Kontrollen, die verwendet werden, um sie zu konstruieren, können Cre-Mäuse so konstruiert werden, dass sie Cre-Rekombinase nur unter bestimmten Bedingungen exprimieren, einschließlich der folgenden: in bestimmten Geweben, wenn die Ernährung einer Maus mit Substanzen wie Doxycyclin, Tetracyclin, RU486 und Tamoxifen ergänzt wird (Brocard et al. 1998; Kellendonk et al. 1999; Utomo et al. 1999) und in bestimmten Entwicklungsstadien.
Abhängig von der Lage und Orientierung der loxP-Stellen in einer Cre-Lox-Maus kann die Cre-Rekombinase Deletionen, Inversionen und Translokationen des floxierten Locus initiieren (Nagy 2000) (Abb. 1).
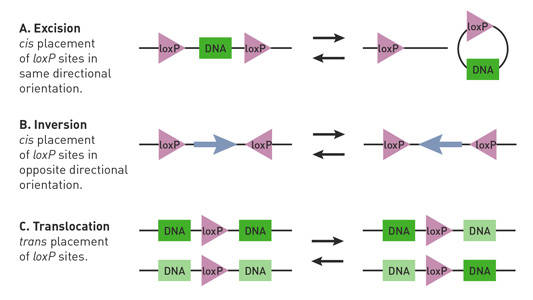 Abb. 1.
Abb. 1.
Cre-Lox-Reaktionen werden durch die Ausrichtung und Lage der loxP-Standorte beeinflusst. Gepaarte loxP-Stellen (Dreiecke) weisen eine Direktionalität auf und können in einer cis- (gleicher DNA-Strang) oder Trans- (verschiedene DNA-Stränge) Anordnung angeordnet sein. (A) Wenn die loxP-Stellen ein DNA-Segment (Rechteck) in einer cis-Anordnung flankieren und in die gleiche Richtung ausgerichtet sind, vermittelt die Cre-Rekombinase die Exzision oder Zirkularisierung des Segments. (B) Wenn die loxP-Stellen das DNA-Segment in einer cis-Anordnung flankieren und in entgegengesetzte Richtungen orientiert sind, vermittelt die Cre-Rekombinase die Inversion des Segments. (C) Wenn sich die loxP-Stellen auf verschiedenen DNA-Strängen befinden und in die gleiche Richtung orientiert sind, vermittelt die Cre-Rekombinase eine Translokation des Segments.
Normalerweise werden Cre- und loxP-Stämme unabhängig voneinander entwickelt und dann gekreuzt. Viele verschiedene Cre-Stämme, die jeweils ein Cre-Transgen unter der Leitung eines anderen gewebespezifischen Promotors enthalten, können mit einem einzigen loxP-Stamm gekreuzt werden. Abhängig davon, welche Stämme gepaart werden, kann eine Vielzahl von Cre-vermittelten Modellsystemen konstruiert werden, einschließlich Transgenen, Knockouts, hypomorphen, reparierbaren Hypomorphen, Chromosomenaberranten und ernährungsinduzierten Mutanten. Durch Mischen und Abgleichen von Cre- und loxP-Stämmen kann ein Forscher die Auswirkungen eines Gens auf gewebespezifische und entwicklungsstadienspezifische Weise untersuchen, die bisher unmöglich waren.
Das FLP-FRT-System
Das FLP-FRT-System ähnelt dem Cre-lox-System und wird in der mausbasierten Forschung immer häufiger eingesetzt. Es geht um die Verwendung von Flippase (FLP) -Rekombinase, abgeleitet von der Hefe Saccharomyces cerevisiae (Sadowski 1995). FLP erkennt ein Paar von FLP-Rekombinase-Target (FRT) -Sequenzen, die eine genomische Region von Interesse flankieren.
Araki K, Imaizumi T, Okuyama K, Oike Y, Yamamura K. 1997. Effizienz der Rekombination durch Cre transiente Expression in embryonalen Stammzellen: Vergleich verschiedener Promotoren. J Biochem (Tokio) 122: 977-82.
Brocard J, Feil R, Chambon P, Metzger D. 1998 Eine chimäre Cre-Rekombinase, die durch synthetische, aber nicht durch natürliche Liganden des Glucocorticoidrezeptors induzierbar ist. Nukleinsäuren Res 26:4086-90.
JAX NOTIZEN. 1999. NIH, Jackson Laboratory und DuPont Pharmaceuticals unterzeichnen Cre-lox Technology Use Agreements. 476:4.
Kellendonk C, Tronche F, Reichardt HM, Schutz G. 1999. Mutagenese des Glukokortikoidrezeptors bei Mäusen. J Steroid Biochem Mol Biol 69:253-9.
Nagy A. 2000. Cre-Rekombinase: das universelle Reagenz für das Genom-Tailoring. Genesis 26:99-109.
Sadowski P. 1995. Die Flp-Rekombinase des 2-µm-Plasmids von Saccharomyces cerevisiae. Prog Nukleinsäure Res Mol Biol 51:53-91.
Sauer B, Henderson N. 1988. Ortsspezifische DNA-Rekombination in Säugetierzellen durch die Cre-Rekombinase des Bakteriophagen P1. In: Proc Natl Acad Sci U S A 85:5166-70.
Sternberg N, Hamilton D. 1981. Bakteriophage P1 ortsspezifische Rekombination. I. Rekombination zwischen loxP-Standorten. J Mol Biol 150:467-86.
Utomo AR, Nikitin AY, Lee WH. 1999. Zeitliche, räumliche und zelltypspezifische Kontrolle der Cre-vermittelten DNA-Rekombination in transgenen Mäusen. Nat Biotechnol 17:1091-6.