Deux des outils génétiques les plus passionnants et polyvalents conçus au cours des 30 dernières années sont les technologies Cre-lox et FLP-FRT. Les deux permettent de réguler étroitement l’emplacement et le moment de l’expression des gènes. Cet article décrit brièvement le fonctionnement des deux systèmes et peut être utilisé dans des modèles de souris.
Le système Cre-lox
La technologie Cre-lox a été introduite dans les années 1980 (Sauer et Henderson 1988; Sternberg et Hamilton 1981) et brevetée par DuPont Pharmaceuticals. Il a été appliqué avec succès dans les levures, les plantes, les cultures de cellules de mammifères et les souris (Araki et al. 1987). Il est basé sur la capacité du gène de recombinase de recombinaison de cyclisation de bactériophage P1 (Cre) à effectuer une recombinaison entre des paires de sites loxP. Une telle recombinaison chez une souris « Cre-lox » (voir ci-dessous) peut activer ou inactiver un gène d’intérêt.
Pour utiliser la technologie Cre-lox, un chercheur doit produire une souris Cre-lox, généralement en élevant une souris Cre à une souris loxP. Une souris Cre contient un transgène de recombinase Cre sous la direction d’un promoteur spécifique au tissu; une souris loxP contient deux sites loxP qui flanquent un segment génomique d’intérêt, le locus « floxed ». En règle générale, les souris Cre et loxP sont produites en utilisant la technologie transgénique (Nagy, 2000). Selon les promoteurs et les autres contrôles réglementaires utilisés pour les construire, les souris Cre ne peuvent être conçues pour exprimer la recombinase Cre que dans certaines conditions, notamment les suivantes: dans certains tissus, lorsque le régime alimentaire d’une souris est complété par des substances telles que la doxycycline, la tétracycline, le RU486 et le tamoxifène (Brocard et al. 1998; Kellendonk et coll. 1999; Utomo et coll. 1999) et à certains stades de développement.
Selon l’emplacement et l’orientation des sites loxP chez une souris Cre-lox, la recombinase Cre peut initier des délétions, des inversions et des translocations du locus floxé (Nagy 2000) (Fig. 1).
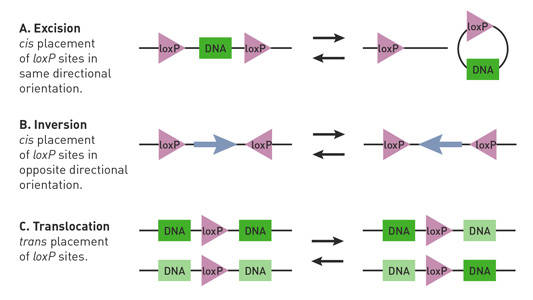 Fig. 1.
Fig. 1.
Les réactions Cre-lox sont affectées par l’orientation et l’emplacement des sites loxP. Les sites loxP appariés (triangles) ont une directivité et peuvent être placés dans un arrangement cis (même brin d’ADN) ou trans (différents brins d’ADN). (A) Si les sites loxP flanquent un segment d’ADN (rectangle) dans un agencement cis et sont orientés dans la même direction, la recombinase Cre médie l’excision ou la circularisation du segment. (B) Si les sites loxP flanquent le segment d’ADN dans un agencement cis et sont orientés dans des directions opposées, la recombinase Cre médie l’inversion du segment. (C) Si les sites loxP sont situés sur des brins d’ADN différents et sont orientés dans la même direction, la recombinase Cre médie une translocation du segment.
Habituellement, les souches Cre et loxP sont développées indépendamment puis croisées. De nombreuses souches de Cre différentes, contenant chacune un transgène de Cre sous la direction d’un promoteur spécifique aux tissus différent, peuvent être croisées avec une seule souche loxP. Selon les souches qui sont accouplées, une variété de systèmes modèles à médiation Cre peuvent être construits, y compris les systèmes transgéniques, les knock-out, les hypomorphes, les hypomorphes réparables, les aberrants chromosomiques et les mutants induits par l’alimentation. En effet, en mélangeant et en faisant correspondre les souches Cre et loxP, un chercheur peut étudier les effets d’un gène de manière spécifique aux tissus et au stade de développement, ce qui était auparavant impossible.
Le système FLP-FRT
Le système FLP-FRT est similaire au système Cre-lox et est de plus en plus fréquemment utilisé dans la recherche sur la souris. Elle implique l’utilisation de la flippase (FLP) recombinase, dérivée de la levure Saccharomyces cerevisiae (Sadowski, 1995). FLP reconnaît une paire de séquences cibles de recombinase FLP (FRT) qui flanquent une région génomique d’intérêt.
Araki K, Imaizumi T, Okuyama K, Oike Y, Yamamura K. 1997. Efficacité de la recombinaison par expression transitoire Cre dans les cellules souches embryonnaires: comparaison de divers promoteurs. J Biochem (Tokyo) 122:977-82.
Brocard J, Feil R, Chambon P, Metzger D. 1998 Une recombinase Cre chimérique inductible par des ligands synthétiques, mais non par des ligands naturels du récepteur des glucocorticoïdes. Acides nucléiques Res 26:4086-90.
NOTES JAX. 1999. NIH, Jackson Laboratory et DuPont Pharmaceuticals signent des accords d’utilisation de la technologie Cre-lox. JAX NOTES 476:4.
Kellendonk C, Tronche F, Reichardt HM, Schutz G. 1999. Mutagenèse du récepteur des glucocorticoïdes chez la souris. J Biochem stéroïde Mol Biol 69: 253-9.
Nagy A. 2000. Recombinase Cre: le réactif universel pour l’adaptation du génome. Genèse 26:99-109.
Sadowski P. 1995. La recombinase Flp du plasmide 2 µm de Saccharomyces cerevisiae. Acide nucléique Prog Res Mol Biol 51:53-91.
Sauer B, Henderson N. 1988. Recombinaison de l’ADN spécifique au site dans les cellules de mammifères par la recombinase Cre du bactériophage P1. Proc Natl Acad Sci U S A 85:5166-70.
Sternberg N, Hamilton D. 1981. Recombinaison spécifique au site du bactériophage P1. I. Recombinaison entre les sites loxP. J Mol Biol 150:467-86.
Utomo AR, Nikitin AY, Lee WH. 1999. Contrôle temporel, spatial et spécifique au type cellulaire de la recombinaison de l’ADN médiée par le Cre chez des souris transgéniques. Nat Biotechnol 17:1091-6.