som MED DNA-isolasjon, stoler forskere ofte PÅ RNA-isolasjonssett for å gjøre livet enklere. Nylig publiserte vi en blogg OM DNA-rensing uten et sett som skisserte flere grunner til at det å gjøre noe uten et sett har fordeler: mindre plastavfall, mindre utgifter og mindre å bli igjen med en rekke tilfeldige løsninger når alle spinnkolonnene går tom. I denne artikkelen dekker vi grunnleggende om å isolere RNA uten et sett.
Tips for å jobbe MED RNA (enten du bruker et sett eller ikke)
Selv om det sier seg selv at man bør ta vare når du gjør NOEN FORM FOR DNA eller RNA rensing for å unngå forurensning, ta ekstra forsiktighet når du utfører RNA-ekstraksjon. RNA er iboende ikke så stabil SOM DNA – det er enkeltstrenget og dets ribosegrupper er utsatt for hydrolyse og varmeforringelse. Videre Er rnaser, eller enzymer som nedbryter RNA, spesielt hardharde proteiner som finnes i og på alt, inkludert huden din. Her er noen generelle tips for å jobbe MED RNA, selv om du bruker et sett:
- bruk alltid hansker, da Rnasene på hendene kan nedbryte RNA.
- Hold et rent arbeidsområde, som kan omfatte sprøyting benken ned med et produkt for å bli kvitt RNases som RNaseZAP.ved høsting av vev, celler, planter, sopp eller bakterier, hold prøvene kalde og arbeid raskt for å redusere rna-nedbrytning.
- Sørg for Å bruke DEPC-behandlet eller RNAse fritt vann. HVIS DU bruker DEPC-behandlet vann, autoklaver vannet for å inaktivere DEPC.
- Kontroller at plast eller glass som brukes Er RNase gratis. RNase-fri plast er lett tilgjengelig fra vitenskapelige leverandører og glass bør behandles MED EN DEPC løsning for 1 time, og autoklaveres for å fjerne rester DEPC. Alternativt kan glassvarer bakes ved 180 hryvnias C i minst 4 timer.
- hvis din SISTE rna-prøve(er) resuspenderes i vann eller te-buffer, må du lagre dem i en -80°c fryser for å forhindre rna-nedbrytning. De vil degradere i en -20°c fryser.
rna ekstraksjonsmetoder utviklet seg til en enkel protokoll som fortsatt brukes i dag
det finnes mange alternative metoder for å isolere DNA uten et sett. Det er imidlertid ikke tilfelle FOR RNA-ekstraksjon og rensing. Det er en enkel metode som fungerer, og variasjoner til den metoden. Et stort hinder for å utvikle protokoller for å isolere RNA var At Rnaser ofte finnes i celler, og UTEN noe å blokkere rnaseaktivitet ved cellelyse, blir RNA degradert. For å effektivt isolere intakt RNA, ville det være nødvendig med en rask, sterk protein denaturant-noe som brøt Ned Rnaser før Rnaser hadde en sjanse til å bryte NED RNA ved cellelyse. på slutten Av 1970-tallet viste Chirgwin og kolleger at et sterkt protein denaturant, guanidiniumtiocyanat, gjorde nettopp dette (Chirgwin et al., 1979). De utviklet en protokoll ment for å isolere RNA fra rotte milter der de homogeniserte milter i en guanidiniumtiocyanatløsning og spunnet ned homogenatet for å fjerne det uoppløselige materialet. Deretter ble homogenatet lastet på cesiumkloridgradienter og ultracentrifugert i opptil 20 timer for å skille det intakte RNA fra DNA og proteiner. Selv om det er svært effektivt å isolere total RNA, krever denne metoden mye TID og avhengig av hvor mange prøver du kan ha, tilgang til en eller flere store, dyre ultracentrifuge.
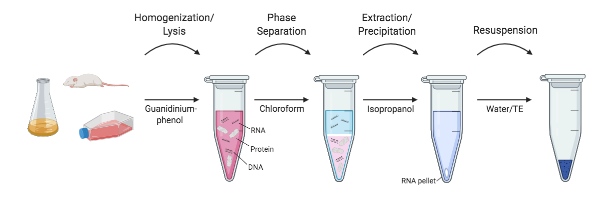
Figur 1: en oversikt over de forskjellige trinnene I RNA-ekstraksjon.Forskere Ved NIH i midten av 1980-tallet satte seg for å utvikle en protokoll som hoppet over ultracentrifugasjonen helt. Chomczynski og Sacchi viste at RNA effektivt kunne separeres fra DNA og proteiner ved en enkel ekstraksjonsprotokoll med guanidiniumtiocyanat-fenol-kloroform. I denne metoden blir prøver fortsatt homogenisert og lysert i en guanidiniumtiocyanatløsning. I stedet for rna-separasjon ved bruk av cesiumkloridgradienter, tilsettes vannmettet fenol, natriumacetat og kloroform til homogenatet og rystes. Etter en rask sentrifugering (ikke ultracentrifugering!), lagene av fenol og kloroform separeres, OG RNA beholdes i det øverste, vandige laget, MENS DNA og andre proteiner beholdes i interfase og bunn, organisk lag. Det øverste vandige laget ekstraheres og RNA kan deretter utfelles isopropanol. Denne metoden reduserte tiden DET tok å isolere RNA fra 20 + timer til rundt 4 timer, og variasjoner på denne no-kit-metoden er fortsatt mye brukt i dag (Chomcynski og Sacchi, 2006).
se vår protokoll for rna-ekstraksjon!
Gjør den enkle protokollen enda mer idiotsikker (fortsatt uten et sett!)
som nevnt ovenfor krever arbeid MED RNA å holde prøvene kalde til homogenisering og cellelyse. Dette kan være utfordrende avhengig av laboratoriesituasjonen eller vevsinnsamlingsmetoden, så bioteknologiselskaper har markedsført flere produkter som bidrar til å effektivisere denne prosessen ytterligere og / eller stabilisere RNA under vevsinnsamling og homogenisering. Den mest kjente av disse produktene er TRIzol® (også kalt Tri Reagent®, Rnazol®, QIAzol® og solgt av mange forskjellige selskaper). TRIzol® er en alt-i-ett syre-guanidinium-fenol-løsning som kombinerer homogeniseringsløsningen og fenoltilsetningen av den opprinnelige no-kit-protokollen i ett trinn. Etter homogenisering i TRIzol® fjernes uoppløselig materiale via sentrifugering, og supernatanten ekstraheres med kloroform som i ovennevnte no-kit-metode. Forskere har også utviklet måter å «stabilisere» RNA i vev før cellelyse. Disse produktene, Nemlig rnalater® fra Termo og RNAProtect® Fra Qiagen, er ammoniumsulfatbaserte løsninger som virker ved å hemme Rnaseaktivitet i celler eller vev – de stabiliserer faktisk ikke rna-molekylene kjemisk (Allewell og Sarma, 1974). I tillegg gir ThermoFisher en protokoll om hvordan Man integrerer rnalater® med Bruk av TRIzol® og ammoniumsulfatstabiliserende løsninger kan gjøres hjemme. et vanlig problem med IKKE-kit rna-ekstraksjonsmetoder er overføring AV DNA som potensielt kan komplisere resultatene av en nedstrøms applikasjon som kvantitativ PCR for å vurdere genuttrykk. Det er flere ting som forskere kan gjøre for å bekjempe dette problemet. Først og fremst, vær oppmerksom på ekstraksjonene dine – hvis du trenger virkelig rent RNA, er det viktig å sørge for at når du trekker ut, bare ta det vandige laget for å unngå overføring AV DNA fra det nederste, organiske laget. Et annet triks er å utfelle RNA ved hjelp av litiumklorid. LiCl-løsninger utfeller selektivt RNA, men IKKE DNA og proteiner. Til slutt, ved Hjelp Av En DNase (det er flere DNase enzym produkter på markedet å velge mellom) på resuspendert rna prøven vil bidra til å sikre DNA-forurensning er ikke et problem.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) Isolering av biologisk aktiv ribonukleinsyre fra kilder beriket i ribonuklease. Biokjemi 18:5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83