duas das ferramentas genéticas mais excitantes e versáteis concebidas nos últimos 30 anos são as tecnologias Cre-lox e FLP-FRT. Ambos permitem que a localização e o momento da expressão do gene sejam regulados de perto. Este artigo descreve brevemente como os dois sistemas funcionam e podem ser usados em modelos de mouse.
o sistema Cre-lox
a tecnologia Cre-lox foi introduzida na década de 1980 (Sauer e Henderson 1988; Sternberg e Hamilton 1981) e patenteada pela DuPont Pharmaceuticals. Foi aplicado com sucesso em leveduras, plantas, culturas celulares de mamíferos e ratos (Araki et al. 1987). Baseia-se na capacidade do gene de recombinação da CRE (bacteriophage cyclization recombination) para efetuar recombinação entre pares de locais de loxP. Tal recombinação em um rato “Cre-lox” (veja abaixo) pode ativar ou inativar um gene de interesse.
para usar a tecnologia Cre-lox, um investigador tem de produzir um rato Cre-lox, tipicamente através da criação de um rato Cre para um rato loxP. Um rato Cre contém um transgeno de recombinase Cre sob a direção de um promotor específico do tecido; um rato loxP contém dois locais loxP que flanqueiam um segmento genômico de interesse, o locus” floxed”. Tipicamente, ratos Cre e loxP são produzidos usando tecnologia transgênica (Nagy 2000). Em função dos promotores e de outros controlos regulamentares utilizados para a sua construção, os ratos Cre só podem ser concebidos para exprimir recombinase Cre em determinadas condições, incluindo:: em certos tecidos, quando a dieta de um rato é complementada com substâncias como a doxiciclina, tetraciclina, RU486 e tamoxifeno (Brocard et al. 1998; Kellendonk et al. 1999; Utomo et al. 1999) e durante certas fases de desenvolvimento. dependendo da localização e orientação dos locais loxP num rato Cre-lox, a recombinase Cre pode iniciar deleções, inversões e translocações do locus floxado (Nagy 2000) (Fig. 1).
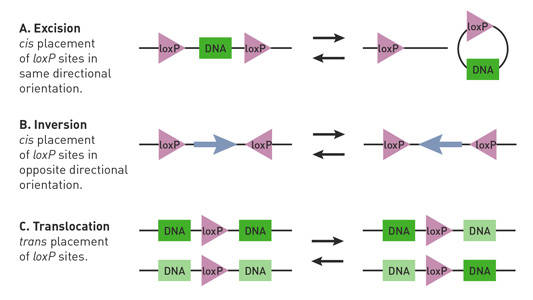 Fig. 1.as reações de Cre-lox são afetadas pela orientação e localização dos locais de loxP. Locais emparelhados de loxP (triângulos) têm direcionalidade e podem ser colocados em um CIS (mesma cadeia de DNA) ou trans (diferentes cadeias de DNA) arranjo. A) se os locais loxP flanqueiam um segmento de ADN (rectângulo) num arranjo cis e se orientam na mesma direcção, a Cre recombinase medeia a excisão ou a circularização do segmento. B) Se os locais loxP flanquearem o segmento de ADN num arranjo cis e forem orientados em direcções opostas, a CRE recombinase mediar a inversão do segmento. C) se os locais loxP estiverem localizados em diferentes cadeias de ADN e forem orientados na mesma direcção, a CRE recombinase mediar uma translocação do segmento.normalmente, as estirpes Cre e loxP são desenvolvidas independentemente e depois cruzadas. Muitas estirpes diferentes de Cre, cada uma contendo um CRE transgene sob a direção de um promotor específico do tecido diferente, podem ser cruzadas com uma única estirpe de loxP. Dependendo de quais estirpes são acasaladas, uma variedade de modelos mediados por Cre podem ser construídos, incluindo transgênicos, knockouts, hipomorfos, repairáveis hipomorfos, aberrações cromossômicas e mutantes induzidos pela dieta. Com efeito, misturando e combinando estirpes Cre e loxP, um investigador pode estudar os efeitos de um gene em formas específicas do tecido e do desenvolvimento que eram anteriormente impossíveis.
Fig. 1.as reações de Cre-lox são afetadas pela orientação e localização dos locais de loxP. Locais emparelhados de loxP (triângulos) têm direcionalidade e podem ser colocados em um CIS (mesma cadeia de DNA) ou trans (diferentes cadeias de DNA) arranjo. A) se os locais loxP flanqueiam um segmento de ADN (rectângulo) num arranjo cis e se orientam na mesma direcção, a Cre recombinase medeia a excisão ou a circularização do segmento. B) Se os locais loxP flanquearem o segmento de ADN num arranjo cis e forem orientados em direcções opostas, a CRE recombinase mediar a inversão do segmento. C) se os locais loxP estiverem localizados em diferentes cadeias de ADN e forem orientados na mesma direcção, a CRE recombinase mediar uma translocação do segmento.normalmente, as estirpes Cre e loxP são desenvolvidas independentemente e depois cruzadas. Muitas estirpes diferentes de Cre, cada uma contendo um CRE transgene sob a direção de um promotor específico do tecido diferente, podem ser cruzadas com uma única estirpe de loxP. Dependendo de quais estirpes são acasaladas, uma variedade de modelos mediados por Cre podem ser construídos, incluindo transgênicos, knockouts, hipomorfos, repairáveis hipomorfos, aberrações cromossômicas e mutantes induzidos pela dieta. Com efeito, misturando e combinando estirpes Cre e loxP, um investigador pode estudar os efeitos de um gene em formas específicas do tecido e do desenvolvimento que eram anteriormente impossíveis.
o sistema FLP-FRT
o sistema FLP-FRT é semelhante ao sistema Cre-lox e está a tornar-se mais frequentemente utilizado na investigação baseada no rato. Envolve a utilização da flippase (FLP) recombinase, derivada da levedura Saccharomyces cerevisiae (Sadowski 1995). FLP reconhece um par de sequências de alvo recombinase FLP (FRT) que flanqueiam uma região genômica de interesse.Araki K, Imaizumi T, Okuyama K,Oike Y, Yamamura K. 1997. Eficiência da recombinação por Cre expressão transitória em células estaminais embrionárias: comparação de vários promotores. J Biochem (Tokyo) 122:977-82.
Brocard J, Feil R, Chambon P, Metzger D. 1998 uma recombinase de Cre quimérica indutível por sintético, mas não por ligantes naturais do receptor glucocorticóide. Ácidos Nucleicos Res 26: 4086-90.JAX NOTES. 1999. A NIH, a Jackson Laboratory e a DuPont Pharmaceuticals assinam acordos de Utilização da tecnologia Cre-lox. JAX NOTA 476: 4.Kellendonk C, Tronche F, Reichardt HM, Schutz G. 1999. Mutagénese do receptor glucocorticóide em ratinhos. J Steroid Biochem Mol Biol 69: 253-9.
Nagy a. 2000. CRE recombinase: the universal reagent for genome tailoring. Genesis 26: 99-109.
Sadowski P. 1995. A recombinase Flp do plasmídeo de 2 µm de Saccharomyces cerevisiae. Ácido Nucleico Prog Res Mol Biol 51: 53-91.
Sauer B, Henderson n. 1988. Recombinação do ADN específico do local nas células de mamíferos pela CRE recombinase do bacteriophage P1. Proc Natl Acad Sci U S A 85: 5166-70.
Sternberg N, Hamilton d. 1981. Recombinação específica do local da bactéria P1. I. recombinação entre locais de loxP. J Mol Biol 150: 467-86.Utomo AR, Nikitin AY, Lee WH. 1999. Controlo específico do tipo Temporal, espacial e celular da recombinação do ADN mediada por Cre em ratinhos transgénicos. Nat Biotechnol 17: 1091-6.