som med DNA-isolering är forskare vanligtvis beroende av RNA-isoleringssatser för att göra livet enklare. Nyligen publicerade vi en blogg om DNA-rening utan ett kit som skisserade flera anledningar till att göra något utan ett kit har fördelar: mindre plastavfall, mindre kostnad och mindre att vara kvar med en massa slumpmässiga lösningar när alla spinnkolumner tar slut. I den här artikeln täcker vi grunderna för att isolera RNA utan ett kit.
Tips för att arbeta med RNA (oavsett om du använder ett kit eller inte)
även om det är självklart att man bör vara försiktig när man gör någon typ av DNA-eller RNA-rening för att undvika kontaminering, var extra försiktig när du utför RNA-extraktion. RNA är i sig inte lika stabilt som DNA – det är enkelsträngat och dess ribosgrupper är mottagliga för hydrolys och värmnedbrytning. Dessutom är Rnaser, eller enzymer som bryter ner RNA, särskilt hårda proteiner som finns i och på allt, inklusive din hud. Här är några allmänna tips för att arbeta med RNA, även om du använder ett kit:
- använd alltid handskar, eftersom Rnaserna på dina händer kan försämra RNA.
- håll ett rent arbetsområde, vilket kan inkludera att spruta din bänk ner med en produkt för att bli av med RNases som RNaseZAP.
- när du skördar vävnader, celler, växter, svampar eller bakterier, håll proverna kalla och arbeta snabbt för att mildra RNA-nedbrytning.
- se till att använda DEPC-behandlat eller RNAse-fritt vatten. Om du använder DEPC-behandlat vatten, autoklavera vattnet för att inaktivera DEPC.
- se till att plastvaror eller glasvaror som används är RNase-fria. RNase-fri plastvaror är lätt tillgänglig från vetenskapliga leverantörer och glasvaror bör behandlas med en DEPC-lösning i 1 timme och autoklaveras för att avlägsna resterande DEPC. Alternativt kan glasvaror bakas vid 180 kcal C i minst 4 timmar.
- Om ditt slutliga RNA-prov(er) resuspenderas i vatten eller te-buffert, förvara dem i en -80 C-Frys för att förhindra RNA-nedbrytning. De kommer att brytas ned i en -20 kcal C Frys.
RNA-extraktionsmetoder utvecklats till ett enkelt protokoll som fortfarande används idag
det finns många alternativa metoder för att isolera DNA utan kit. Det är emellertid inte fallet för RNA-extraktion och rening. Det finns en enkel metod som fungerar, och variationer till den metoden. Ett stort hinder för att utveckla protokoll för att isolera RNA var att Rnaser ofta finns i celler, och utan något att blockera Rnasaktivitet vid celllys bryts RNA ned. För att effektivt isolera intakt RNA skulle en snabb, stark proteindenaturant krävas-något som bröt ner Rnaser innan Rnaser hade en chans att bryta ner RNA vid celllys.
i slutet av 1970-talet visade Chirgwin och kollegor att en stark proteindenaturant, guanidiniumtiocyanat, gjorde just detta (Chirgwin et al., 1979). De utvecklade ett protokoll som var avsett för att isolera RNA från råtta mjältar där de homogeniserade mjältar i en guanidiniumtiocyanatlösning och snurrade ner homogenatet för att avlägsna det olösliga materialet. Sedan laddades homogenatet på cesiumkloridgradienter och ultracentrifugerades i upp till 20 timmar för att separera det intakta RNA från DNA och proteiner. Även om det är mycket effektivt att isolera totalt RNA, kräver denna metod mycket tid och beroende på hur många prover du kan ha, tillgång till en eller flera stora, dyra ultracentrifuge.
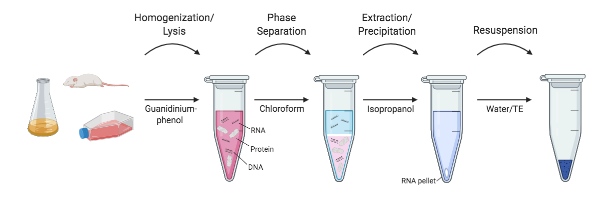
Figur 1: en översikt över de olika stegen i RNA-extraktion.
forskare vid NIH i mitten av 1980-talet bestämde sig för att utveckla ett protokoll som hoppade över ultracentrifugationen helt och hållet. Chomczynski och Sacchi visade att RNA effektivt kunde separeras från DNA och proteiner genom ett enkelt extraktionsprotokoll med guanidiniumtiocyanat-fenolkloroform. I denna metod homogeniseras prover fortfarande och lyseras i en guanidiniumtiocyanatlösning. I stället för RNA-separation med användning av cesiumkloridgradienter tillsätts emellertid vattenmättad fenol, natriumacetat och kloroform till homogenatet och skakas. Efter en snabb centrifugering (inte ultracentrifugering!), skikten av fenol och kloroform separeras, och RNA behålls i det övre, vattenhaltiga skiktet, medan DNA och andra proteiner behålls i interfasen och botten, organiskt skikt. Det övre vattenhaltiga skiktet extraheras och RNA kan sedan utfällas isopropanol. Denna metod minskade tiden det tog att isolera RNA från 20+ timmar till cirka 4 timmar, och variationer på denna no-kit-metod används fortfarande i stor utsträckning idag (Chomcynski och Sacchi, 2006).
Visa vårt protokoll för RNA-extraktion!
gör det enkla protokollet ännu mer idiotsäkert (fortfarande utan kit!)
som nämnts ovan kräver arbete med RNA att dina prover hålls kalla tills homogenisering och celllys. Detta kan vara utmanande beroende på din laboratoriesituation eller vävnadssamlingsmetod, så bioteknikföretag har marknadsfört flera produkter som hjälper till att ytterligare effektivisera denna process och/eller stabilisera RNA under vävnadssamling och homogenisering. Den mest kända av dessa produkter är TRIzol (även kallad Tri reagens, Rnazol, QIAzol och säljs av många olika företag). TRIzol är en allt-i-ett-syra-guanidinium-fenol-lösning som kombinerar homogeniseringslösningen och fenoltillsatsen av det ursprungliga no-kit-protokollet i ett steg. Efter homogenisering i trizol, avlägsnas olösligt material via centrifugering och supernatanten extraheras med kloroform som i ovanstående no-kit-metod.
forskare har också utvecklat sätt att” stabilisera ” RNA i vävnader före celllys. Dessa produkter, nämligen rnalater 2G från Thermo och rnaprotect 2G från Qiagen, är ammoniumsulfatbaserade lösningar som fungerar genom att hämma RNAs – aktivitet i celler eller vävnader-de stabiliserar inte kemiskt RNA-molekylerna (Allewell och Sarma, 1974). Dessutom, ThermoFisher ger ett protokoll om hur man kan integrera rnalater vanilj med användning av trizol och ammoniumsulfat stabiliserande lösningar kan göras i huset.
ett vanligt problem med No-kit RNA-extraktionsmetoder är överföring av DNA som potentiellt kan komplicera resultat av en nedströms applikation såsom kvantitativ PCR för att bedöma genuttryck. Det finns flera saker som forskare kan göra för att bekämpa denna fråga. Först och främst, var uppmärksam på dina extraktioner – om du behöver riktigt rent RNA är det viktigt att se till att när du extraherar, att bara ta det vattenhaltiga skiktet för att undvika överföring av DNA från botten, organiskt lager. Ett annat knep är att fälla ut RNA med litiumklorid. LiCl-lösningar fäller selektivt RNA, men inte DNA och proteiner. Slutligen, med hjälp av ett DNase (det finns flera DNase-enzymprodukter på marknaden att välja mellan) på ditt resuspenderade RNA-prov kommer det att bidra till att DNA-kontaminering inte är ett problem.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) isolering av biologiskt aktiv ribonukleinsyra från källor berikade i ribonukleas. Biokemi 18:5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83