Wie bei der DNA-Isolierung verlassen sich Wissenschaftler häufig auf RNA-Isolations-Kits, um ihr Leben zu erleichtern. Kürzlich haben wir einen Blog über DNA-Reinigung ohne Kit veröffentlicht, in dem mehrere Gründe dargelegt wurden, warum etwas ohne Kit Vorteile hat: weniger Plastikmüll, weniger Kosten und weniger die Möglichkeit, mit einer Reihe zufälliger Lösungen zurückgelassen zu werden, wenn alle Spin-Säulen leer sind. In diesem Artikel behandeln wir die Grundlagen der Isolierung von RNA ohne Kit.
Tipps für die Arbeit mit RNA (unabhängig davon, ob Sie ein Kit verwenden oder nicht)
Obwohl es selbstverständlich ist, dass man bei jeder Art von DNA- oder RNA-Reinigung vorsichtig sein sollte, um eine Kontamination zu vermeiden, sollten Sie bei der RNA-Extraktion besonders vorsichtig sein. RNA ist von Natur aus nicht so stabil wie DNA – sie ist einzelsträngig und ihre Ribosegruppen sind anfällig für Hydrolyse und Wärmeabbau. Darüber hinaus sind RNasen oder Enzyme, die RNA abbauen, besonders robuste Proteine, die in und auf allem, einschließlich Ihrer Haut, vorkommen. Hier sind einige allgemeine Tipps für die Arbeit mit RNA, auch wenn Sie ein Kit verwenden:
- Tragen Sie immer Handschuhe, da die RNasen an Ihren Händen RNA abbauen können.Halten Sie einen sauberen Arbeitsbereich, der das Besprühen Ihrer Bank mit einem Produkt einschließen kann, um RNases wie RNaseZAP loszuwerden.
- Wenn Sie Gewebe, Zellen, Pflanzen, Pilze oder Bakterien ernten, halten Sie die Proben kalt und arbeiten Sie schnell, um den RNA-Abbau zu mildern.
- Achten Sie darauf, DEPC-behandeltes oder RNase-freies Wasser zu verwenden. Wenn Sie DEPC-behandeltes Wasser verwenden, autoklavieren Sie das Wasser, um das DEPC zu inaktivieren.
- Stellen Sie sicher, dass verwendete Kunststoff- oder Glaswaren RNase-frei sind. Glaswaren sollten 1 Stunde lang mit einer DEPC-Lösung behandelt und autoklaviert werden, um restliches DEPC zu entfernen. Alternativ können Glaswaren mindestens 4 Stunden bei 180 ° C gebacken werden.
- Wenn Ihre endgültige(n) RNA-Probe(n) in Wasser oder TE-Puffer resuspendiert werden, lagern Sie sie in einem Gefrierschrank bei -80°C, um einen RNA-Abbau zu verhindern. Sie werden in einem -20 ° C Gefrierschrank abgebaut.
RNA-Extraktionsmethoden entwickelten sich zu einem einfachen Protokoll, das heute noch verwendet wird
Es gibt viele alternative Methoden zur Isolierung von DNA ohne Kit. Dies ist jedoch bei der RNA-Extraktion und -reinigung nicht der Fall. Es gibt eine einfache Methode, die funktioniert, und Variationen dieser Methode. Eine große Hürde bei der Entwicklung von Protokollen zur Isolierung von RNA war, dass RNasen häufig in Zellen vorkommen und ohne etwas, das die RNase-Aktivität bei der Zelllyse blockiert, RNA abgebaut wird. Um intakte RNA effektiv zu isolieren, wäre ein schneller, starker Proteindenaturierer erforderlich – etwas, das RNasen abbaut, bevor RNasen eine Chance hatten, RNA bei der Zelllyse abzubauen. In den späten 1970er Jahren zeigten Chirgwin und Kollegen, dass ein starker Proteindenaturierer, Guanidiniumthiocyanat, genau dies tat (Chirgwin et al., 1979). Sie entwickelten ein Protokoll zur Isolierung von RNA aus Rattenmilzen, bei dem sie Milzen in einer Guanidiniumthiocyanatlösung homogenisierten und das Homogenat herunterschleuderten, um das unlösliche Material zu entfernen. Dann wurde das Homogenat auf Cäsiumchlorid-Gradienten geladen und bis zu 20 Stunden ultrazentrifugt, um die intakte RNA von DNA und Proteinen zu trennen. Obwohl diese Methode bei der Isolierung von Gesamt-RNA sehr effektiv ist, erfordert sie viel Zeit und je nachdem, wie viele Proben Sie haben, Zugang zu einer oder mehreren großen, teuren Ultrazentrifugen.
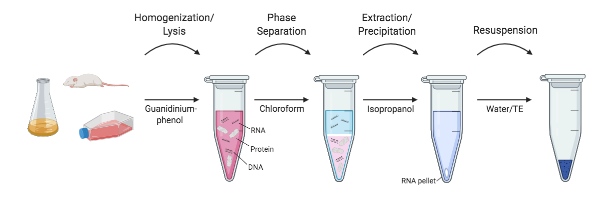
Abbildung 1: Ein Überblick über die verschiedenen Schritte bei der RNA-Extraktion.
Mitte der 1980er Jahre machten sich Forscher am NIH daran, ein Protokoll zu entwickeln, das die Ultrazentrifugation insgesamt übersprang. Chomczynski und Sacchi zeigten, dass RNA durch ein einfaches Extraktionsprotokoll mit Guanidiniumthiocyanat-Phenol-Chloroform effektiv von DNA und Proteinen getrennt werden kann. Bei dieser Methode werden die Proben noch homogenisiert und in einer Guanidiniumthiocyanatlösung lysiert. Anstelle einer RNA-Trennung mit Cäsiumchlorid-Gradienten werden dem Homogenat jedoch wassergesättigtes Phenol, Natriumacetat und Chloroform zugesetzt und geschüttelt. Nach einer schnellen Zentrifugation (nicht Ultrazentrifugation!), trennen sich die Phenol- und Chloroformschichten, und RNA wird in der oberen wässrigen Schicht zurückgehalten, während DNA und andere Proteine in der Interphase und der unteren organischen Schicht zurückgehalten werden. Die obere wässrige Schicht wird extrahiert und RNA kann dann Isopropanol ausgefällt werden. Diese Methode reduzierte die Zeit, die benötigt wurde, um RNA von 20+ Stunden auf etwa 4 Stunden zu isolieren, und Variationen dieser No-Kit-Methode sind heute noch weit verbreitet (Chomcynski und Sacchi, 2006).
Sehen Sie sich unser Protokoll für die RNA-Extraktion an!
Das einfache Protokoll noch narrensicherer machen (noch ohne Kit!)
Wie oben erwähnt, erfordert die Arbeit mit RNA, dass Ihre Proben bis zur Homogenisierung und Zelllyse kalt gehalten werden. Daher haben Biotechnologieunternehmen mehrere Produkte auf den Markt gebracht, die dazu beitragen, diesen Prozess weiter zu rationalisieren und / oder die RNA während der Gewebeentnahme und -homogenisierung zu stabilisieren. Das bekannteste dieser Produkte ist TRIzol® (auch TRI Reagent®, RNAzol®, QIAzol® genannt und von vielen verschiedenen Unternehmen vertrieben). TRIzol® ist eine All-in-One-Säure-Guanidinium-Phenol-Lösung, die die Homogenisierungslösung und die Phenolzugabe des ursprünglichen No-Kit-Protokolls in einem Schritt kombiniert. Nach Homogenisierung in TRIzol® wird unlösliches Material durch Zentrifugation entfernt und der Überstand wie in der obigen No-Kit-Methode mit Chloroform extrahiert. Forscher haben auch Wege entwickelt, RNA in Geweben vor der Zelllyse zu „stabilisieren“. Diese Produkte, nämlich RNAlater® von Thermo und RNAProtect® von Qiagen, sind Lösungen auf Ammoniumsulfatbasis, die die RNase-Aktivität in Zellen oder Geweben hemmen – sie stabilisieren die RNA-Moleküle nicht chemisch (Allewell und Sarma, 1974). Darüber hinaus bietet ThermoFisher ein Protokoll zur Integration von RNAlater® in die Verwendung von TRIzol® und Ammoniumsulfat-Stabilisierungslösungen können im eigenen Haus hergestellt werden. Ein häufiges Problem bei No-Kit-RNA-Extraktionsmethoden ist die Verschleppung von DNA, die möglicherweise die Ergebnisse einer nachgelagerten Anwendung wie der quantitativen PCR zur Beurteilung der Genexpression erschweren kann. Es gibt mehrere Dinge, die Forscher tun können, um dieses Problem zu bekämpfen. Achten Sie in erster Linie auf Ihre Extraktionen – wenn Sie wirklich saubere RNA benötigen, ist es wichtig, dass Sie beim Extrahieren nur die wässrige Schicht nehmen, um eine Verschleppung der DNA von der unteren, organischen Schicht zu vermeiden. Ein weiterer Trick besteht darin, die RNA mit Lithiumchlorid auszufällen. LiCl-Lösungen fällen selektiv RNA, aber nicht DNA und Proteine. Schließlich hilft die Verwendung einer DNase (es gibt mehrere DNase-Enzymprodukte auf dem Markt zur Auswahl) auf Ihrer resuspendierten RNA-Probe sicherzustellen, dass eine DNA-Kontamination kein Problem darstellt.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) Isolierung biologisch aktiver Ribonukleinsäure aus mit Ribonuklease angereicherten Quellen. Biochemie 18: 5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83