kuten DNA-eristyksessä, tutkijat luottavat yleensä RNA-eristyspakkauksiin helpottaakseen elämäänsä. Äskettäin julkaisimme blogin DNA: n puhdistuksesta ilman pakkausta, jossa hahmoteltiin useita syitä siihen, miksi ilman pakkausta tekemisellä on etuja: vähemmän muovijätettä, vähemmän kuluja ja vähemmän sattumanvaraisia ratkaisuja, kun kaikki spin-sarakkeet loppuvat. Tässä artikkelissa käsitellään RNA: n eristämisen perusteita ilman pakkausta.
vinkkejä RNA: n kanssa työskentelyyn (käytitpä pakkausta tai et)
vaikka on sanomattakin selvää, että on oltava varovainen kaikissa DNA-tai RNA-puhdistuksissa kontaminaation välttämiseksi, on erityisen varovainen RNA: n uuttamisessa. RNA ei ole luonnostaan yhtä stabiili kuin DNA-se on yksijuosteinen ja sen riboosiryhmät ovat herkkiä hydrolyysille ja lämmön hajoamiselle. Lisäksi Rnaasit eli RNA: ta hajottavat entsyymit ovat erityisen sitkeitä proteiineja, joita on kaikessa, myös ihossasi. Tässä muutamia yleisiä vinkkejä RNA: n kanssa työskentelyyn, vaikka käyttäisitkin pakkausta:
- käytä aina hanskoja, sillä käsissäsi olevat Rnaasit voivat hajottaa RNA: ta.
- pidä puhdas työalue, johon voi kuulua penkkisi suihkuttaminen alas tuotteella Rnaasien, kuten Rnasezapin, poistamiseksi.
- kerätessään kudoksia, soluja, kasveja, sieniä tai bakteereja, pitää näytteet kylminä ja vaikuttaa nopeasti RNA: n hajoamisen hillitsemiseksi.
- varmista, että käytät DEPC-käsiteltyä tai Rnaasivapaata vettä. Jos käytät DEPC-käsiteltyä vettä, autoklaavoi vesi DEPC: n inaktivoimiseksi.
- varmista, että käytetyt muovi-tai lasiesineet ovat Rnaasivapaita. Rnaasivapaita muoviesineitä on helposti saatavilla tieteellisiltä toimittajilta ja lasiesineet on käsiteltävä DEPC-liuoksella 1 tunnin ajan ja autoklavoitava depc-jäännöksen poistamiseksi. Vaihtoehtoisesti lasiesineitä voidaan paistaa 180°C: ssa vähintään 4 tuntia.
- Jos lopullinen RNA-näytteesi sekoitetaan uudelleen veteen tai TE-puskuriin, säilytä ne -80°C: n pakastimessa RNA: n hajoamisen estämiseksi. Ne hajoavat -20°C: n pakkasessa.
RNA: n uuttamismenetelmät kehittyivät yksinkertaiseksi protokollaksi, jota käytetään vielä nykyäänkin
on olemassa monia vaihtoehtoisia menetelmiä DNA: n eristämiseksi ilman koesarjaa. Näin ei kuitenkaan ole RNA: n uuttamisessa ja puhdistamisessa. On yksi yksinkertainen menetelmä, joka toimii, ja muunnelmia, että menetelmä. Merkittävä este kehittää protokollia eristää RNA oli, että rnaasit ovat yleisesti löytyy soluissa, ja ilman jotain estää RNaasi toimintaa, kun solu lysis, RNA hajoaa. Ehjän RNA: n tehokkaaseen eristämiseen tarvittaisiin nopea, vahva proteiinin denaturointiaine – jotain, joka hajotti Rnaasit ennen kuin rnaasit ehtivät hajottaa RNA: ta solujen hajoamisen yhteydessä.
1970-luvun lopulla Chirgwin kollegoineen osoitti, että vahva proteiinidenaturantti, guanidiniumtiosyanaatti, teki juuri tämän (Chirgwin et al., 1979). He kehittivät RNA: n eristämiseen rotan pernoista tarkoitetun protokollan, jossa he homogenoivat pernat guanidiniumtiosyanaattiliuoksessa ja kehräsivät homogenaatin alas liukenemattoman aineen poistamiseksi. Sitten homogenaatti lastattiin cesium-kloridigladientteihin ja ultracentrifugoitiin jopa 20 tunnin ajan, jotta ehjä RNA erotettiin DNA: sta ja proteiineista. Vaikka tämä menetelmä on erittäin tehokas koko RNA: n eristämisessä, se vaatii paljon aikaa ja riippuen siitä, kuinka monta näytettä sinulla voi olla, pääsyn yhteen tai useampaan suureen, kalliiseen ultracentrifugiin.
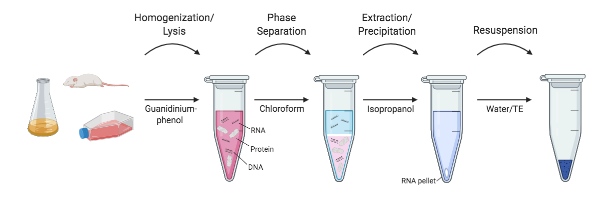
kuva 1: hahmotelma RNA-uuttamisen eri vaiheista.
NIH: n tutkijat lähtivät 1980-luvun puolivälissä kehittämään protokollaa, joka ohitti ultracentrifugaation kokonaan. Chomczynski ja Sacchi osoittivat, että RNA voitiin erottaa tehokkaasti DNA: sta ja proteiineista yksinkertaisella uuttoprotokollalla guanidiniumtiosyanaatti-fenoli-kloroformilla. Tässä menetelmässä näytteet vielä homogenoidaan ja lysytetään guanidiniumtiosyanaattiliuoksessa. Sen sijaan, että RNA erotettaisiin cesium-kloridigladienteillä, homogenaattiin lisätään vesikyllästettyä fenolia, natriumasetaattia ja kloroformia ja ravistetaan. Nopean sentrifugoinnin jälkeen (ei ultracentrifugointia!), fenoli-ja kloroformikerrokset eroavat toisistaan ja RNA säilyy ylimmässä, vesipitoisessa kerroksessa, kun taas DNA ja muut proteiinit säilyvät interfaasi-ja alimmassa, orgaanisessa kerroksessa. Ylin vesipitoinen kerros uutetaan ja RNA voidaan sitten saostaa isopropanoliksi. Tämä menetelmä lyhensi RNA: n eristämiseen kulunutta aikaa 20+ tunnista noin 4 tuntiin, ja tämän No-kit-menetelmän muunnelmia käytetään edelleen laajalti (Chomcynski and Sacchi, 2006).
Katso protokollamme RNA: n poistoon!
tekee yksinkertaisesta protokollasta vielä idioottivarmemman (vielä ilman pakkia!)
kuten edellä mainittiin, RNA: n kanssa työskentely edellyttää näytteiden pitämistä kylminä homogenisaatioon ja solujen hajoamiseen asti. Tämä voi olla haastavaa riippuen laboratoriotilanteesta tai kudoskeräysmenetelmästä, joten biotekniikkayritykset ovat markkinoineet useita tuotteita, jotka auttavat edelleen virtaviivaistamaan tätä prosessia ja/tai vakauttamaan RNA: ta kudoskeräyksen ja homogenoinnin aikana. Tunnetuin näistä tuotteista on TRIzol® (kutsutaan myös nimellä TRI Reagent®, RNAzol®, QIAzol® ja sitä myyvät monet eri yritykset). TRIzol® on all-in-one happo-guanidinium-fenoliliuos, joka yhdistää alkuperäisen no-kit-protokollan homogenointiliuoksen ja fenolin lisäämisen yhteen vaiheeseen. Trizol® – homogenoinnin jälkeen liukenematon materiaali poistetaan sentrifugoimalla ja supernatantti uutetaan kloroformilla kuten edellä mainitussa no-kit-menetelmässä.
tutkijat ovat myös kehittäneet tapoja ”vakauttaa” RNA kudosten sisällä ennen solujen hajoamista. Nämä tuotteet, nimittäin Thermon rnalater® ja QIAGENin RNAProtect®, ovat ammoniumsulfaattipohjaisia ratkaisuja, jotka toimivat estämällä rnaasiaktiivisuutta soluissa tai kudoksissa – ne eivät oikeastaan vakauta kemiallisesti RNA-molekyylejä (Allewell and Sarma, 1974). Lisäksi Lämpökalastaja tarjoaa protokollan siitä, miten integroida Rnalater® käyttämällä TRIzol® ja ammoniumsulfaatin stabilointiliuokset voidaan tehdä talossa.
yleinen ongelma no-kit-RNA-uuttomenetelmissä on DNA: n siirtyminen, joka voi mahdollisesti vaikeuttaa tuloksia, jotka saadaan kvantitatiivisesta PCR: stä geeniekspression arviointiin. On olemassa useita asioita, että tutkijat voivat tehdä tämän ongelman torjumiseksi. Ennen kaikkea, ole tietoinen uuttamisestasi – jos tarvitset todella puhdasta RNA: ta, on tärkeää varmistaa, että uuttaessasi otat vain vesipitoisen kerroksen välttääksesi DNA: n kulkeutumisen alhaalta, orgaanisesta kerroksesta. Toinen kikka on saostaa RNA litiumkloridin avulla. LiCl-liuokset saostavat selektiivisesti RNA: ta, mutta eivät DNA: ta ja proteiineja. Lopuksi käyttämällä dnase (on olemassa useita dnase entsyymi tuotteita markkinoilla valita) teidän uudelleen suspendoitu RNA näyte auttaa varmistamaan DNA saastuminen ei ole ongelma.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) biologisesti aktiivisen ribonukleiinihapon eristäminen ribonukleaasilla rikastetuista lähteistä. Biokemia 18: 5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83