Come con l’isolamento del DNA, gli scienziati comunemente si affidano a kit di isolamento di RNA per rendere la loro vita più facile. Recentemente, abbiamo pubblicato un blog sulla purificazione del DNA senza kit che ha delineato diversi motivi per cui fare qualcosa senza kit ha dei vantaggi: meno rifiuti di plastica, meno spese e meno di essere lasciati con un mucchio di soluzioni casuali quando tutte le colonne di spin si esauriscono. In questo articolo, copriamo le basi dell’isolamento dell’RNA senza un kit.
Suggerimenti per lavorare con l’RNA (se si utilizza un kit o meno)
Anche se va da sé che si dovrebbe fare attenzione ogni volta che si fa qualsiasi tipo di DNA o RNA purificazione per evitare la contaminazione, prestare particolare attenzione quando si esegue l’estrazione di RNA. L’RNA non è intrinsecamente stabile quanto il DNA – è singolo incagliato ed i suoi gruppi del ribosio sono suscettibili di idrolisi e di degradazione del calore. Inoltre, gli RNASI, o enzimi che degradano l’RNA, sono proteine particolarmente resistenti che si trovano in e su tutto, compresa la pelle. Ecco alcuni consigli generali per lavorare con l’RNA, anche se si utilizza un kit:
- Indossare sempre i guanti, poiché gli RNA sulle mani possono degradare l’RNA.
- Mantenere un’area di lavoro pulita, che può includere spruzzare il banco con un prodotto per sbarazzarsi di RNasi come RNaseZAP.
- Quando si raccolgono tessuti, cellule, piante, funghi o batteri, tenere i campioni freddi e lavorare rapidamente per mitigare la degradazione dell’RNA.
- Assicurarsi di utilizzare acqua trattata DEPC o priva di RNasi. Se si utilizza acqua trattata DEPC, autoclave l’acqua per inattivare il DEPC.
- Assicurarsi che i prodotti in plastica o in vetro utilizzati siano privi di RNasi. La plastica priva di RNase è prontamente disponibile da fornitori scientifici e la vetreria deve essere trattata con una soluzione DEPC per 1 ora e sterilizzata in autoclave per rimuovere il DEPC residuo. In alternativa, la vetreria può essere cotta a 180°C per almeno 4 ore.
- Se i campioni finali di RNA vengono risospesi in acqua o tampone TE, conservarli in un congelatore a -80°C per prevenire la degradazione dell’RNA. Si degradano in un congelatore a -20°C.
I metodi di estrazione dell’RNA si sono evoluti in un semplice protocollo ancora usato oggi
Esistono molti metodi alternativi per isolare il DNA senza un kit. Tuttavia, questo non è il caso per l’estrazione e la purificazione dell’RNA. C’è un metodo semplice che funziona e variazioni a quel metodo. Un ostacolo importante per lo sviluppo di protocolli per isolare l’RNA era che le RNasi si trovano comunemente nelle cellule, e senza qualcosa per bloccare l’attività della RNasi sulla lisi cellulare, l’RNA viene degradato. Per isolare efficacemente l’RNA intatto, sarebbe necessario un denaturante proteico veloce e forte, qualcosa che scomponesse gli RNASI prima che gli RNasi avessero la possibilità di scomporre l’RNA dopo la lisi cellulare.
Alla fine del 1970, Chirgwin e colleghi hanno dimostrato che un forte denaturante proteico, il tiocianato di guanidinio, ha fatto proprio questo (Chirgwin et al., 1979). Hanno sviluppato un protocollo pensato per isolare l’RNA dagli spleens di ratto in cui hanno omogeneizzato gli spleens in una soluzione di tiocianato di guanidinio e hanno filato l’omogenato per rimuovere il materiale insolubile. Quindi, l’omogenato è stato caricato su gradienti di cloruro di cesio e ultracentrifugato per un massimo di 20 ore per separare l’RNA intatto dal DNA e dalle proteine. Anche se molto efficace nell’isolare l’RNA totale, questo metodo richiede molto tempo e, a seconda di quanti campioni si possono avere, l’accesso a uno o più grandi, costosi ultracentrifuge.
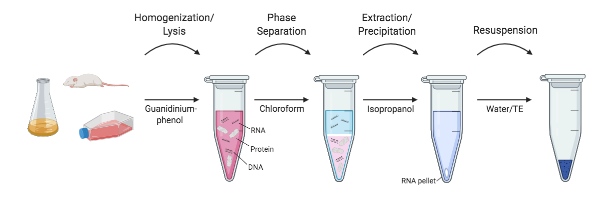
Figura 1: Una descrizione delle diverse fasi di estrazione dell’RNA.
I ricercatori del NIH a metà degli anni 1980 hanno deciso di sviluppare un protocollo che ha saltato del tutto l’ultracentrifugazione. Chomczynski e Sacchi hanno dimostrato che l’RNA potrebbe essere efficacemente separato dal DNA e dalle proteine mediante un semplice protocollo di estrazione con tiocianato di guanidinio-fenolo-cloroformio. In questo metodo, i campioni sono ancora omogeneizzati e lisati in una soluzione di tiocianato di guanidinio. Tuttavia, invece della separazione dell’RNA usando gradienti di cloruro di cesio, il fenolo saturo d’acqua, l’acetato di sodio e il cloroformio vengono aggiunti all’omogenato e scossi. Dopo una rapida centrifugazione (non ultracentrifugazione!), gli strati di fenolo e cloroformio si separano e l’RNA viene trattenuto nello strato acquoso superiore, mentre il DNA e altre proteine vengono trattenuti nell’interfase e nello strato organico inferiore. Lo strato acquoso superiore viene estratto e l’RNA può quindi essere precipitato isopropanolo. Questo metodo ha ridotto il tempo necessario per isolare l’RNA da 20+ ore a circa 4 ore, e le variazioni su questo metodo no-kit sono ancora ampiamente utilizzate oggi (Chomcynski e Sacchi, 2006).
Visualizza il nostro protocollo per l’estrazione dell’RNA!
Rendere il protocollo simple ancora più infallibile (ancora senza kit!)
Come accennato in precedenza, lavorare con l’RNA richiede di mantenere i campioni freddi fino all’omogeneizzazione e alla lisi cellulare. Questo può essere difficile a seconda della situazione di laboratorio o del metodo di raccolta dei tessuti, quindi le aziende biotecnologiche hanno commercializzato diversi prodotti che aiutano a semplificare ulteriormente questo processo e/o stabilizzare l’RNA durante la raccolta e l’omogeneizzazione dei tessuti. Il più noto di questi prodotti è TRIzol® (chiamato anche TRI Reagent®, RNAzol®, QIAzol® e venduto da molte aziende diverse). TRIzol® è una soluzione all-in-one acido-guanidinio-fenolo che combina la soluzione di omogeneizzazione e l’aggiunta di fenolo del protocollo originale no-kit in un unico passaggio. Dopo l’omogeneizzazione in TRIzol®, il materiale insolubile viene rimosso tramite centrifugazione e il surnatante viene estratto con cloroformio come nel metodo di cui sopra no-kit.
I ricercatori hanno anche sviluppato modi per” stabilizzare ” l’RNA all’interno dei tessuti prima della lisi cellulare. Questi prodotti, vale a dire RNAlater® di Thermo e RNAProtect® di Qiagen, sono soluzioni a base di solfato di ammonio che agiscono inibendo l’attività della RNasi all’interno delle cellule o dei tessuti-in realtà non stabilizzano chimicamente le molecole di RNA (Allewell e Sarma, 1974). Inoltre, ThermoFisher fornisce un protocollo su come integrare RNAlater® con l’uso di TRIzol® e soluzioni stabilizzanti di solfato di ammonio può essere fatto in casa.
Un problema comune con i metodi di estrazione dell’RNA senza kit è il riporto del DNA che può potenzialmente complicare i risultati di un’applicazione a valle come la PCR quantitativa per valutare l’espressione genica. Ci sono diverse cose che i ricercatori possono fare per combattere questo problema. Prima di tutto, essere consapevoli delle vostre estrazioni – se avete bisogno di RNA veramente pulito, è importante assicurarsi che quando si estrae, prendere solo lo strato acquoso per evitare riporto di DNA dal fondo, strato organico. Un altro trucco è quello di precipitare l’RNA utilizzando cloruro di litio. Le soluzioni di LiCl precipitano selettivamente l’RNA, ma non il DNA e le proteine. Infine, utilizzando una DNasi (ci sono diversi prodotti enzimatici DNasi sul mercato tra cui scegliere) sul campione di RNA risospeso contribuirà a garantire la contaminazione del DNA non è un problema.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) Isolamento di acido ribonucleico biologicamente attivo da fonti arricchite in ribonucleasi. Biochimica 18: 5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83