zoals bij DNA-isolatie, vertrouwen wetenschappers vaak op RNA-isolatiekits om hun leven gemakkelijker te maken. Onlangs publiceerden we een blog over DNA purification without a kit die verschillende redenen schetste waarom iets doen zonder een kit voordelen heeft: minder plastic afval, minder kosten, en minder van het worden overgelaten met een bos van willekeurige oplossingen wanneer alle spin kolommen opraken. In dit artikel behandelen we de basisprincipes van het isoleren van RNA zonder kit.
Tips voor het werken met RNA (of u nu een kit gebruikt of niet)
hoewel het vanzelfsprekend is dat men voorzichtig moet zijn bij het uitvoeren van een DNA-of RNA-zuivering om contaminatie te voorkomen, wees extra voorzichtig bij het uitvoeren van RNA-extractie. RNA is inherent niet zo stabiel zoals DNA-het is vastgelopen en zijn ribosegroepen zijn vatbaar voor hydrolyse en hittedegradatie. Bovendien zijn RNases, of enzymen die RNA afbreken, vooral sterke eiwitten die in en op alles worden gevonden, inclusief je huid. Hier zijn enkele algemene tips voor het werken met RNA, zelfs als u een kit gebruikt:
- draag altijd handschoenen, omdat de RNases op uw handen RNA kunnen degraderen.
- Houd een schoon werkgebied, waaronder het bespuiten van uw bank met een product om RNases zoals RNaseZAP te verwijderen.
- bij het oogsten van weefsels, cellen, planten, schimmels of bacteriën, monsters koud houden en snel werken om de afbraak van RNA te verminderen.
- zorg ervoor dat u DEPC-behandeld of RNAse-vrij water gebruikt. Indien met DEPC behandeld water wordt gebruikt, moet het water in de autoclaaf worden geplaatst om de DEPC te inactiveren.
- zorg ervoor dat gebruikt plasticgerei of glaswerk RNase vrij is. RNase-vrij plasticware is direct verkrijgbaar bij wetenschappelijke leveranciers en glaswerk moet worden behandeld met een DEPC-oplossing voor 1 uur, en autoclaaf om resterende DEPC verwijderen. Als alternatief kan glaswerk minimaal 4 uur bij 180°C worden gebakken.
- als uw uiteindelijke RNA-monster(s) worden geresuspendeerd in water of te-buffer, bewaar deze dan in een vriezer van -80°C om degradatie van RNA te voorkomen. Ze zullen degraderen in een -20°C vriezer.
RNA-extractiemethoden evolueerden tot een eenvoudig protocol dat nog steeds wordt gebruikt
Er zijn veel alternatieve methoden om DNA zonder kit te isoleren. Dat is echter niet het geval voor RNA-extractie en-zuivering. Er is een eenvoudige methode die werkt, en variaties op die methode. Een belangrijke hindernis aan het ontwikkelen van protocollen om RNA te isoleren was dat RNases algemeen in cellen worden gevonden, en zonder iets om RNase-activiteit op cellysis te blokkeren, wordt RNA gedegradeerd. Om intact RNA effectief te isoleren, zou een snel, sterk eiwit denaturant nodig zijn-iets dat rnases afbrak voordat RNases een kans hadden om RNA op cellysis af te breken. aan het eind van de jaren zeventig toonden Chirgwin en zijn collega ‘ s aan dat een sterk proteïne denaturant, guanidiniumthiocyanaat, juist dit deed (Chirgwin et al., 1979). Ze ontwikkelden een protocol bedoeld voor het isoleren van RNA van rat milten waarin ze gehomogeniseerd milten in een guanidinium thiocyanaat oplossing en gesponnen naar beneden het homogenaat om het onoplosbare materiaal te verwijderen. Vervolgens werd het homogenaat op cesium-chloride gradiënten geladen en tot 20 uur ultracentrifugeerd om het intacte RNA van DNA en eiwitten te scheiden. Hoewel zeer effectief in het isoleren van totaal RNA, vereist deze methode veel tijd en afhankelijk van hoeveel monsters je kan hebben, toegang tot een of meer grote, dure ultracentrifuge.
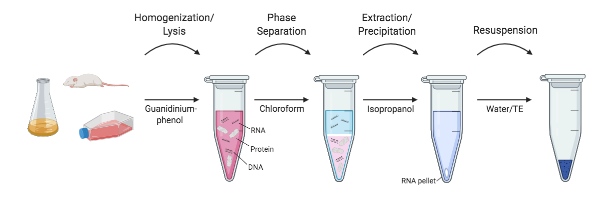
figuur 1: een overzicht van de verschillende stappen in RNA-extractie.
onderzoekers aan de NIH in het midden van de jaren 1980 begonnen met het ontwikkelen van een protocol dat de ultracentrifugatie helemaal oversloeg. Chomczynski en Sacchi toonden aan dat RNA effectief kon worden gescheiden van DNA en eiwitten door een eenvoudig extractieprotocol met guanidinium thiocyanaat-fenol-chloroform. Bij deze methode worden de monsters nog steeds gehomogeniseerd en gelyseerd in een guanidiniumthiocyanaatoplossing. Echter, in plaats van RNA-scheiding met behulp van cesium-chloride gradiënten, water-verzadigde fenol, natriumacetaat, en chloroform worden toegevoegd aan het homogenaat en geschud. Na een snelle centrifugering (niet ultracentrifugering!), scheiden de lagen van fenol en chloroform, en RNA wordt behouden in de hoogste, waterige laag, terwijl DNA en andere proteã nen in de interfase en bodem, organische laag worden behouden. De bovenste waterige laag wordt geëxtraheerd en RNA kan dan isopropanol worden neergeslagen. Deze methode verminderde de tijd die het kostte om RNA van 20+ uren te isoleren tot rond 4 uren, en variaties op deze geen-kit methode worden nog wijd gebruikt vandaag (Chomcynski en Sacchi, 2006).
Bekijk ons protocol voor RNA-extractie!
maakt het eenvoudige protocol nog onfeilbaarder (nog steeds zonder kit!)
zoals hierboven vermeld, vereist het werken met RNA het koud houden van uw monsters tot homogenisatie en cellysis. Dit kan een uitdaging zijn, afhankelijk van uw laboratoriumsituatie of weefselinzamelingsmethode, dus biotechnologiebedrijven hebben verschillende producten op de markt gebracht die helpen om dit proces verder te stroomlijnen en/of RNA te stabiliseren tijdens weefselinzameling en homogenisatie. De meest bekende van deze producten is TRIzol® (ook wel TRI reagens®, RNAzol®, QIAzol® genoemd en verkocht door veel verschillende bedrijven). TRIzol® is een alles-in-één zuur-guanidinium-fenoloplossing die de homogenisatieoplossing en de fenoltoevoeging van het oorspronkelijke no-kit-protocol in één stap combineert. Na homogenisatie in TRIzol® wordt het onoplosbare materiaal via centrifugering verwijderd en wordt het supernatant met chloroform geëxtraheerd, zoals bij de bovenstaande no-kit-methode.
onderzoekers hebben ook manieren ontwikkeld om RNA in weefsels te” stabiliseren ” vóór de lysis van de cellen. Deze producten, namelijk RNAlater ® van Thermo en RNAProtect® van Qiagen, zijn op ammoniumsulfaat gebaseerde oplossingen die werken door RNase – activiteit in cellen of weefsels te remmen-ze stabiliseren de RNA-moleculen eigenlijk niet chemisch (Allewell en Sarma, 1974). Daarnaast biedt ThermoFisher een protocol voor de integratie van RNAlater® met het gebruik van TRIzol® en kunnen ammoniumsulfaat stabiliserende oplossingen in eigen huis worden gemaakt.
een veel voorkomend probleem met No-kit RNA-extractiemethoden is de overdracht van DNA die mogelijk de resultaten van een downstreamtoepassing, zoals kwantitatieve PCR, kan compliceren om de genexpressie te beoordelen. Er zijn verschillende dingen die onderzoekers kunnen doen om dit probleem te bestrijden. Wees eerst en vooral bewust van je extracties – als je echt schoon RNA nodig hebt, is het belangrijk om ervoor te zorgen dat bij het extraheren, alleen de waterige laag te nemen om overdracht van DNA van de bodem, organische laag te voorkomen. Een andere truc is om het RNA te precipiteren met behulp van lithiumchloride. LiCl-oplossingen precipiteren selectief RNA, maar niet DNA en eiwitten. Tot slot, het gebruik van een DNase (er zijn verschillende DNase enzymproducten op de markt om uit te kiezen) op uw geresuspendeerde RNA-monster zal helpen ervoor te zorgen DNA-besmetting is geen probleem.

Chirgwin JM, Przybyla AE, MacDonald RJ, Rutter WJ (1979) isolatie van biologisch actief ribonucleïnezuur uit bronnen verrijkt met ribonuclease. Biochemistry 18: 5294-5299. https://doi.org/10.1021/bi00591a005
Chomczynski P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate–phenol–chloroform extraction: twenty-something years on. Nature Protocols 1:581–585. https://doi.org/10.1038/nprot.2006.83