dwa najbardziej ekscytujące i wszechstronne narzędzia genetyczne zaprojektowane w ciągu ostatnich 30 lat to technologie Cre-lox i FLP-FRT. Oba pozwalają na ścisłą regulację lokalizacji i czasu ekspresji genów. W tym artykule krótko opisano, w jaki sposób oba systemy działają i mogą być używane w modelach myszy.
system Cre-lox
technologia Cre-lox została wprowadzona w latach 80. (Sauer and Henderson 1988; Sternberg and Hamilton 1981) i opatentowana przez firmę DuPont Pharmaceuticals. Jest z powodzeniem stosowany w drożdżach, roślinach, hodowlach komórek ssaków i myszach(Araki et al. 1987). Opiera się on na zdolności genu rekombinacji cyklicznej P1 bakteriofagów (Cre) do rekombinacji między parami miejsc loxP. Taka rekombinacja u myszy „Cre-lox” (patrz poniżej) może aktywować lub inaktywować interesujący Gen.
aby użyć technologii Cre-lox, badacz musi wyprodukować mysz Cre-lox, zazwyczaj poprzez hodowlę myszy Cre do myszy loxP. Mysz Cre zawiera transgen rekombinazy Cre pod kierunkiem promotora tkankowo-specyficznego; mysz loxP zawiera dwa miejsca loxP, które flankują interesujący segment genomowy, locus” floxed”. Zazwyczaj myszy Cre i loxP są produkowane przy użyciu technologii transgenicznej (Nagy 2000). W zależności od promotorów i innych kontroli regulacyjnych użytych do ich konstruowania, myszy Cre mogą być zaprojektowane do ekspresji rekombinazy Cre tylko pod pewnymi warunkami, w tym pod następującymi warunkami: w niektórych tkankach, gdy dieta myszy jest uzupełniana substancjami takimi jak doksycyklina, tetracyklina, RU486 i tamoksyfen (Brocard et al. 1998; Kellendonk et al. 1999; Utomo et al. 1999) oraz w niektórych stadiach rozwojowych.
w zależności od lokalizacji i orientacji miejsc loxP u myszy Cre-lox, rekombinaza Cre może inicjować delecje, inwersje i translokacje flokowanego locus (Nagy 2000) (Fig. 1).
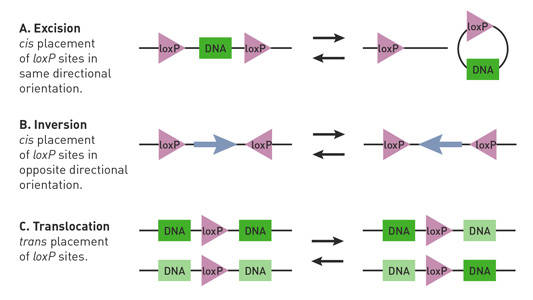 1.
1.
na reakcje Cre-lox wpływa Orientacja i lokalizacja miejsc loxP. Sparowane miejsca loxP (Trójkąty) mają kierunkowość i mogą być umieszczone w układzie cis (ta sama nić DNA) lub trans (różne nici DNA). (A) jeśli miejsca loxP flankują segment DNA (prostokąt) w układzie cis i są zorientowane w tym samym kierunku, rekombinaza Cream pośredniczy w wycięciu lub okrężnym wycięciu segmentu. B) Jeśli miejsca loxP flankują segment DNA w układzie cis i są zorientowane w przeciwnych kierunkach, rekombinaza Cre pośredniczy w inwersji segmentu. C) Jeśli miejsca loxP znajdują się na różnych nici DNA i są zorientowane w tym samym kierunku, rekombinaza Cre pośredniczy w translokacji segmentu.
Zwykle szczepy Cre i loxP rozwijają się niezależnie, a następnie krzyżują. Wiele różnych szczepów Cre, z których każdy zawiera transgen Cre pod kierunkiem innego promotora tkankowo-specyficznego, można krzyżować z jednym szczepem loxP. W zależności od tego, które szczepy są połączone, można skonstruować różne systemy modelowe, w tym transgeniki, knockouty, hipomorfy, naprawialne hipomorfy, aberranty chromosomowe i mutanty indukowane dietą. W efekcie, mieszając i dopasowując szczepy Cre i loxP, badacz może badać wpływ genu w specyficzny dla tkanek i stadium rozwoju sposób, który wcześniej był niemożliwy.
system FLP-FRT
System FLP-FRT jest podobny do systemu Cre-lox i jest coraz częściej wykorzystywany w badaniach opartych na myszach. Polega na zastosowaniu rekombinazy flippazy (FLP) pochodzącej z drożdży Saccharomyces cerevisiae (Sadowski 1995). FLP rozpoznaje parę sekwencji docelowych rekombinazy FLP (FRT), które flankują interesujący region genomowy.
Araki K, Imaizumi T, Okuyama K, Oike Y, Yamamura K. 1997. Efektywność rekombinacji przez ekspresję Kredytywną w embrionalnych komórkach macierzystych: porównanie różnych promotorów. J Biochem (Tokyo) 122: 977-82.
Brocard J, Feil R, Chambon P, Metzger D. 1998 a chimeric Cre recombinase inducible by synthetic, but not by natural ligands of the glikokortykosteroid receptor. Nucleic Acids Res 26: 4086-90.
JAX NOTES. 1999. NIH, Jackson Laboratory i DuPont Pharmaceuticals podpisują umowy o wykorzystaniu technologii Cre-lox. JAX NOTES 476: 4.
Kellendonk C, Tronche F, Reichardt HM, Schutz G. 1999. Mutageneza receptora glukokortykoidowego u myszy. J Steroid Biochem Mol Biol 69: 253-9.
Nagy A. 2000. Cre recombinase: uniwersalny odczynnik do dostosowywania genomu. Księga Rodzaju 26: 99-109.
Sadowski P. 1995. Rekombinaza Flp 2-µm plazmidu Saccharomyces cerevisiae. Prog Nucleic Acid Res Mol Biol 51: 53-91.
Sauer B, Henderson N. 1988. Miejscowa rekombinacja DNA w komórkach ssaków przez rekombinazę Cre bakteriofaga P1. Proc Natl Acad Sci U S A 85: 5166-70.
Sternberg N, Hamilton D. 1981. Rekombinacja specyficzna dla miejsca P1 bakteriofagów. I. rekombinacja między miejscami loxP. J Mol Biol 150:467-86.
Utomo AR, Nikitin AY, Lee WH. 1999. Czasowa, przestrzenna i specyficzna dla typu komórki Kontrola rekombinacji DNA za pośrednictwem Cre u myszy transgenicznych. Nat Biotechnol 17: 1091-6.