A Microbial Biorealm page on the genus Citrobacter freundii
Classification
Higher order taxa
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Citrobacter
Species
NCBI: Taxonomy
Citrobacter Freundii
Description and significance
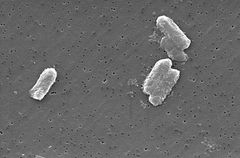 Photo: Pete Wardell / CDC
Photo: Pete Wardell / CDC
gatunki Citrobacter, w tym Citrobacter freundii, są tlenowymi pałeczkami gram-ujemnymi. Citrobacter freundii to bakterie o długich pręcikach, zwykle o długości 1-5 µm . Większość komórek C. freundii jest otoczona przez wiele wici używanych do poruszania się, ale kilka z nich nie jest ruchliwych. Jego siedlisko obejmuje środowisko (gleba, woda, ścieki), żywność i drogi jelitowe zwierząt i ludzi . Należy do rodziny Enterobacteriaceae.
jako patogen oportunistyczny, C. freundii jest odpowiedzialny za szereg istotnych zakażeń oportunistycznych. Wiadomo, że jest przyczyną różnych zakażeń szpitalnych dróg oddechowych, dróg moczowych, krwi i kilku innych normalnie sterylnych miejsc u pacjentów . C. freundii stanowi około 29% wszystkich zakażeń oportunistycznych . Dlatego jednym z głównych powodów sekwencjonowania wielu różnych szczepów i plazmidów genomu C. freundii jest znalezienie antybiotyków, które mogą zwalczać te oportunistyczne infekcje.
Co zaskakujące, ten zakaźny drobnoustrój u ludzi odgrywa pozytywną rolę w środowisku. C. freundii odpowiada za redukcję azotanów do azotynów w środowisku . Ta kluczowa konwersja jest ważnym etapem w cyklu azotowym. Recykling azotu jest bardzo istotny, ponieważ atmosfera ziemska to około 85% azotu . Dlatego też, ze względu na jego istotny wkład w środowisko jest kolejną motywacją do sekwencjonowania genomu C. freundii.
rodzaj Citrobacter został odkryty w 1932 roku przez Werkmana i Gillena. Kultury C. freundii zostały wyizolowane i zidentyfikowane w tym samym roku z ekstraktów glebowych .
struktura genomu
Brak informacji o kompletnym genomie C. freundii jest dostępnych online, chociaż niektóre pojedyncze szczepy i plazmidy mikroorganizmów zostały zsekwencjonowane. Najbardziej znanym z nich jest plazmid pCTX-m#3, ponieważ jest największym plazmidem i koduje dużą ilość białek. Jej kolejność została zakończona 6 stycznia 2005 roku. Jest to okrągły plazmid DNA o długości 89 468 par zasad nukleotydowych. Długość plazmidu wynosi 0,089468 (Mbp). Składa się z 51,0% zawartości GC i koduje 105 białek .
kolejna ważna cecha C. Genom freundii polega na tym, że jest to jedyny mikrob w rodzinie Enterobacteriaceae, który zawiera plazmid kodujący γ-liazę L-metioniny (MGL). Sekwencja nukleotydowa plazmidu zawiera wstawkę EcoRI o długości 3000 bp . Fragment zawiera również dwie otwarte ramki do czytania. Pierwsza ramka składa się z 1194 nukleotydów, a druga-1296 nukleotydów.Pierwsza ramka, znana jako gen megL, koduje białko 398 reszt aminokwasowych, które ma homologię sekwencji z MGL z różnych źródeł. Druga ramka koduje białko o homologii sekwencji z białkami należącymi do rodziny permeaz .
sekwencjonowano również gen β-laktamazy C. freundii OS60 AmpC, który składa się ze 1197 nukleotydów. Koduje on prekursor o długości 380 aminokwasów i zawiera peptyd sygnałowy o długości 19 reszt na końcu 5′. Gen ten koduje Dojrzałe białko o masie cząsteczkowej 39 781 Daltonów. Pozycje aminokwasów w tych prekursorach są suprisinlgy identyczne z pozostałościami w chromosomowych ampc β-laktamazach E. coli K12.
kolejny ważny szczep w genomie C. freunii jest GN346, który jest izolatem klinicznym odzyskanym w 1965 roku. Szczep ten wytwarza enzym cefalosporynazę, który ma zdolność hydrolizowania i inaktywacji cefalosporyn anibitoinowych i cefamycyny . Regiony strukturalne i promotorowe genu cefalosporynazy mają długość 1408 nukleotydów. Sekwencja aminokwasowa dojrzałego enzymu składa się z 361 aminokwasów o masie cząsteczkowej 39 878 Da .
struktura komórkowa i metabolizm
struktura komórkowa C. freundi jest długa i pręcikowata, zwykle o długości 1-5 µm. Na zewnątrz komórki znajduje się wiele wici wykorzystywanych do motalności . Ponieważ C. freundii jest bakterią gram-ujemną, zawiera dwie błony (wewnętrzną i zewnętrzną).Przestrzeń peryplazmiczna znajduje się pomiędzy dwoma błonami. Błona zewnętrzna nie zawiera źródła energii, ale zawiera wiele Porin osadzonych w środku, które pomagają organizmowi zdobyć ważne jony . W przeciwieństwie do bakterii gram-dodatnich, komórki C. freundii nie zawierają grubej ściany komórkowej złożonej z peptydoglikanu.
dla metabolizmu, C. freundii ma niesamowitą zdolność do wzrostu na glicerolu jako jedynym źródłem węgla i energii. W tym procesie glicerol jest fermentowany w procesie dysmutacji. Proces ten wymaga dwóch ścieżek . W pierwszym szlaku glicerol jest dehydrogenowany przez dehydrogenazę glicerolu związaną z NAD1 do dihydroksyacetonu. Dihydroksyaceton jest następnie fosforylowany i doprowadzany do glikolizy przez kinazę dihydroksyacetonową . W drugim szlaku glicerol jest odwodniony przez dehydratazę glicerolu zależną od koenzymu B12, tworząc aldehyd 3-hydroksypropionowy . Produkt ten jest redukowany do głównego produktu fermentacji 1,3-propanodiolu przez związaną z nadh dehydrogenazę 1,3-propanodiolu, która regeneruje NAD1. Regulon DHA koduje cztery niezbędne enzymy tych dwóch szlaków. Co zadziwiające, ekspresja regulonu dha jest indukowana tylko wtedy, gdy obecny jest glicerol .
komórki C. freundii są również zdolne do metabolizowania laktozy lub cytrynianu jako źródła węgla .
Ekologia
Citrobacter freundii występuje powszechnie w środowisku, głównie w glebie, wodzie i kanalizacji. Są one wskaźnikiem potencjalnego zanieczyszczenia wody. Występują również na różnych narządach chorych zwierząt, w tym ssaków, ptaków, gadów i płazów . Nie wiadomo, czy wchodzą w interakcje z innymi organimami.
w środowisku C. freundii może przekształcić azotan lub jon amonowy (który jest atomem azotu połączonym z czterema atomami wodoru) w azotyn; reakcja ta zachodzi w środowisku, a także w przewodzie pokarmowym ludzi i innych zwierząt . Po przekształceniu azotanu w azotyn w środowisku, azotyn jest przekształcany w azot, a ten ostatni etap kończy cykl azotowy w atmosferze Ziemi, która składa się z 85% azotu . Ekologiczna rola tego organizmu obejmuje nie tylko jego ważną rolę w cyklu azotowym, ponieważ może również gromadzić uran (który jest podstawowym materiałem w technologii jądrowej) poprzez budowanie kompleksów fosforanowych .
Citrobacter freundii był również badany pod kątem biodegradacji kwasu garbnikowego stosowanego w garbnikach .
patologia
jako patogen oportunistyczny, Citrobacter freundii jest często przyczyną znaczących zakażeń oportunistycznych, co oznacza, że na ogół nie powoduje chorób u zdrowych ludzkich gospodarzy. Dotyczą tylko pacjentów ze słabym układem odpornościowym, co oznacza, że potrzebują „szansy” na zarażenie osoby . Dlatego u pacjentów z osłabionym układem odpornościowym znane są gatunki Cytrobacter powodujące wiele różnych zakażeń szpitalnych dróg oddechowych, dróg moczowych i krwi . Choroby wątroby, dróg żółciowych i trzustki są również powszechnymi chorobami wywoływanymi przez C. freundii. Dróg żółciowych jest najczęstszym miejscem zakażenia przez prątki C. freundii.
jedną śmiertelną chorobą, z którą C. freundii był związany, jest noworodkowe zapalenie opon mózgowo-rdzeniowych. Noworodkowe zapalenie opon mózgowo-rdzeniowych jest zapalenie opon mózgowo-rdzeniowych (system błon otaczających OUN) z powodu inwazji bakterii . Śmiertelność w przypadku zapalenia opon mózgowo-rdzeniowych Citrobacter jest niedopuszczalnie wysoka, a śmiertelność pacjentów wynosi od 25 do 50 %. Ponadto, poważne problemy neurologiczne nadal utrzymują się u 75% osób, które przeżyły. W tej chorobie Citrobacter freundii jest w stanie przeniknąć przez barierę krew-mózg, która składa się z nabłonka splotu naczyniówkowego i śródbłonka naczyń włosowatych mózgu .
badania przeprowadzone przez Badger et. al w artykule „Citrobacter freundii atakuje i replikuje w ludzkich mózgowych mikronaczyniowych komórkach śródbłonka” sugeruje, że proliferacja bakterii C. freundii odbywa się na poziomie wewnątrzkomórkowym, co było sprzeczne z ogólną myślą naukową. Odkrycia wskazują, że C. freundii przemieszcza się przez wakuole, replikuje się i jest uwalniany do bazolateralnej strony ludzkich komórek śródbłonka mózgu (hbmec) w celu przekroczenia bariery krew-mózg. Dalsza analiza może potencjalnie pozwolić na strategie terapeutyczne w leczeniu zakażeń. Nadal nie ma możliwości leczenia.
niektóre choroby badane u pstrąga i cyprynidów są również wywoływane przez C. freundii. C. freundii powoduje nieprawidłowe zmiany zapalne w jelitach pstrąga oraz zmiany zapalne i martwicze w narządach wewnętrznych karpiowatych. Choroba została odkryta za pomocą sztucznej infekcji czystą kulturą C. freundii. Odkrycie to ustaliło, że C. freundii jest przyczyną chorób ryb .
w badaniu przypadku przeprowadzonym przez Journal of Medical Microbiology, u pacjenta rozwinęło się zapalenie otrzewnej i zakażenie tunelowe z powodu Citrobacter freundii, które jest rzadkie. Pacjent był poddawany ciągłej ambulatoryjnej dializie otrzewnowej. Zwykle czynnikami powodującymi są mikroorganizmy gram-dodatnie, w szczególności Staphylococcus aureus i staphyloccus edpidermis. Nie są również znane doniesienia o infekcji tunelowej spowodowanej przez C. freundii. Wstępna antybiotykoterapia nie zadziałała i infekcja utrzymywała się do czasu usunięcia cewnika. Jest to istotne klinicznie, ponieważ lek Citrobacter Freundii wykazuje różną wrażliwość na antybiotyki, dlatego początkowe leczenie nie zakończyło się sukcesem. Pacjent nie reagował na leczenie, dopóki nie usunięto cewnika, pokazując, że Citrobacter freundii to patogeny oportunistyczne, które wpływają na pacjentów hospitalizowanych i z obniżoną odpornością .
zastosowanie w biotechnologii
w przemyśle biotechnologicznym Citrobacter freundii wytwarza wiele ważnych enzymów.Pierwsze enzymy, które wytwarza, to fosfataza. Aktywność fosfatazy C. freundii postulowano udział w akumulacji ołowiu, który może odegrać ważną rolę w przemyśle biotechnologicznym. Odkryto również, że aktywność fosfatazy C. freundii ma odporność na niektóre Odczynniki diagnostyczne .
Oczyszczanie i charakterystyka bakteryjnej beta-liazy selenocysteiny, enzymu, który specyficznie katalizuje rozszczepienie l-selenocysteiny do L-alaniny, zostało oczyszczone z Citrobacter freundii. Enzym jest monomeryczny o masie cząsteczkowej ok. 64 000 i zawiera 1 mol 5 ’ – fosforanu pirydoksalu jako kofaktora na mol enzymu. Enzym katalizuje również alfa, beta eliminację beta-chloro-L-alaniny do postaci NH3, pirogronianu .
szczepy C. Freundii posiadają również plazmid kodujący cefalosporynazę AmpC klasy 1. Enzymy te mogą hydrolizować inaktywować nowe cefamycyny i cefalosporyny .
aktualne badania
badania na małą skalę dotyczące niektórych szczepów C. freundii przeprowadzono niedawno na University of Tennessee, Knoxville. Znaczenie niektórych genów oporności na tetracykliny i streptomycynę oraz integronów klasy 1 w C. oceniano izolację freundii z gleb gospodarstw mlecznych i gleb niewysokich. Jeden szczep C. freundii wyekstrahowany z gleb gospodarstw mlecznych nosił integrony klasy 1 z różnymi wstawionymi kasetami genów. Wyniki tego małego badania sugerują, że obecność wielu genów oporności i integronów klasy 1 W C. freundii w glebie gospodarstwa mlecznego może działać jako rezerwuar genów oporności przeciwdrobnoustrojowej i może odgrywać rolę w rozpowszechnianiu tych genów oporności przeciwdrobnoustrojowej do innych komensalnych i rdzennych społeczności drobnoustrojów w glebie. Konieczne są jednak dodatkowe, długoterminowe badania prowadzone w większej liczbie lokalizacji, aby wesprzeć tę hipotezę .
przeprowadzono drugie badania nad C. freundii w celu opracowania metody reakcji łańcuchowej polimerazy (PCR), która jednocześnie wykorzystuje trzy pary specyficznych starterów do wykrywania genów niektórych drobnoustrojów (w tym C. freundii). Metoda obejmowała zaprojektowanie trzech par starterów, którymi były: SPVC-1 i SPVC-2, INVA-1 i INVA – 2 oraz VIAB-1 i VIAB-2. PCR przeprowadzono przy użyciu tych trzech starterów do identyfikacji 14 klinicznie ważnych organizmów bakteryjnych. Następujące szczepy zostały szybko zidentyfikowane za pomocą PCR: (1) C. freundii; (2) s. Typhi; i S. Paratyphi C; (3) s. Dublin (antygen wirulencji-dodatni); i (4) serowary Salmonella, które zawierają plazmid wirulencji typu spv. Chociaż ta metoda PCR jest nowa, wraz z postępem technologicznym w przyszłości metoda ta może umożliwić natychmiastową identyfikację C. freundii u ssaków, tak aby można było bezzwłocznie rozpocząć odpowiednie leczenie antybiotykami .
trzecie badanie dotyczące C. freundiego zostało przeprowadzone na Uniwersytecie w Barcelonie w Hiszpanii. Badano mechanizmy oporności na fluorochinolony u dwóch szczepów Citrobacter freundii. Oba szczepy wyizolowano od tego samego pacjenta. Badania te pozwoliły na częściową charakterystykę genów acrA i acrB tego mikroorganizmu. Ekspresję genów w obu szczepach analizowano za pomocą mikromacierzy DNA dla Escherichia coli. Podobieństwo nukleotydów między częściowo zsekwencjonowanymi genami acrA i acrB C. freundii i E. coli wynosiło odpowiednio 80,7% i 85%. Geny acrA i acrB C. freundii są podobne do genów E. coli i ich nadekspresja mogą odgrywać ważną rolę w modulowaniu ostatecznego minimalnego stężenia fluorochinolonów hamującego .
czwarte badanie dotyczące C. freundii przeprowadzono na Tajwanie. Zespół naukowców wyizolował pacjenta z cukrzycą, u którego rozwinęło się martwicze zapalenie powięzi spowodowane przez C. freundii w wyniku urazu wywołanego przez zwierzę morskie. Martwicze zapalenie powięzi jest zakażeniem w głębszej warstwie skóry i tkanek podskórnych. Podczas leczenia pacjenta pobrano próbkę płynu z rany i znaleziono C. freundii. Po trzech dniach od rozpoczęcia leczenia antybiotykami cefotaksminą i cefepminą doszło do nagromadzenia ropni podskórnych. Po 6, 10, 14 i 21 dniach opieki nad pacjentem, niektóre antybiotyki dawały pacjentowi ulgę, ale nie osiągnięto długotrwałego powrotu do zdrowia. Cefotoaksym, cefepim, ciprofloksamina należą do antybiotyków, które nie pasowały do C. freundii. Pacjent w pełni wyzdrowiał po 42 dniach leczenia ertapenem. Naukowcy wyizolowali dwie kolonie C. freundii w ciągu 5 dni od siebie. W miarę wzrostu izolatów stały się one niezwykle odporne na cefotaksym i cefepim. Powodem, dla którego entrapenem działał przeciwko C. freundii jest to, że jest aktywny przeciwko AmpC wytwarzającym Enterobacteriaceae .
oporność na antybiotyki
gatunki Cytrobakterów są częstą przyczyną zakażeń szpitalnych związanych z pacjentami poddawanymi długotrwałemu leczeniu szpitalnemu. Ostatnio doniesiono, że C. freundii wykazuje oporność na antybiotyki o szerokim spektrum działania, w tym piperacyl, piperacylintazobaktam, wankomycynę i cefalosporyny. Izolacja opornego na ceftriakson Citrobacter freundii (CRCF) była związana z nadmiernie przepisywanymi antybiotykami o szerokim spektrum działania. Pojawiające się nowe szczepy CRCF mogą sugerować indukcję lub depresję genów oporności, jak również eliminację konkurujących ze sobą organizmów. CRCF był głównie izolowany od pacjentów z istotnymi chorobami współistniejącymi, w tym AIDS, chorobą naczyń obwodowych i chorobą naczyń mózgowych. Stwierdzono również, że stosowanie fluorochinolonu nie ma wpływu na izolację CRCF .
wiadomo, że Citrobacter freundii zawiera w swoim chromosomie gen kodujący cefalosporynazę. Enzym ten hydrolizuje Wiązanie−CO-NH w pierścieniu laktamowym cefalosporyn i cephamyci, czyniąc bakterie odpornymi na tego typu antybiotyki. Jednak po wystawieniu na nowe cefemy i cerbapenemy trzeciej generacji, klinicznie wyizolowany C. freundii wykazywał wrażliwość na te substancje.Na oddziale chirurgicznym szpitala uniwersyteckiego w Nagoi u pacjentów poddanych zabiegom chirurgicznym zaobserwowano niewielki wybuch C. freundii oporny na cefemy trzeciej generacji. C. freundii izolowano z żółci pacjenta, gazy ranowej, kału, ropy i wodobrzusza. Zasugerowano, że te nowe szczepy C. freundii zawierały plazmid kodujący AmpC celfalosporynazę, ale po braku przeniesienia oporności cefemów z C. freundii na E. coli stwierdzono, że enzym musi być kodowany w chromosomie C. freundii. Ponieważ C. freundi jest związane z zakażeniami szpitalnymi, zaleca się ostrożność w stosunku do tych nowych szczepów .
1. Wang JT, Chang SC, Chen YC, Luh KT. „Porównanie wrażliwości antybakteryjnej izolatów Citrobacter freundii w dwóch różnych okresach czasu.”The Journal of Microbiology, Immunology and Infection. 2000 Dec; 33(4): 258-62.
2. Whalen JG, Mully TW, Enlgish JC 3rd. „Spontaniczne zakażenie Cytrobacter freundii u immunokompetentnego pacjenta.”Archiwum dermatologii. 2007 Jan; 143(1): 124-5.
3. Puchenkowa SG. „Enterobacteria in areas of water along the Crimean Coast.”Mikrobiolohichnyĭ zhurnal. 1996 Marzec-Kwiecień; 58 (2): 3-7.
4.http://www.ncbi.nlm.nih.gov/sites/entrez?db=genomeprj&cmd=Retrieve&dopt=Overview&list_uids=13123
5. Ilya V. Manukhov, Daria V. Mamaeva, Sergei M. Rastorguev, Nicolai G. Faleev, Elena A. Morozova, Tatyana V. Demidkina, and Gennadii B. Zavilgelsky. „Gen kodujący γ-Liazę L-metioniny jest obecny w genomach rodziny Enterobacteriaceae: identyfikacja i charakterystyka γ-liazy L-metioniny Cytrobacter freundii.”The Journal of Bacteriology. 2005 June; 187(11): 3889-3893.
6. Lindberg, Frederik; Westman, Lennart; Normark, Staffan. „Składniki regulacyjne W indukcji β-laktamazy Citrobacter freundii ampC.”Proceedings of the National Academy of Sciences of the United States of America. 1985 lipiec; 82(14): 4620-4624.
7. Tsukamoto K, Tachibana K, Yamazaki N, Ishii Y, Ujiie K, Nishida N, Sawai T. ” rola lizyny-67 w miejscu aktywnym beta-laktamazy klasy C z Citrobacter freundii GN346.”European Journal of Biochemistry / FEBS. 1990 Feb 22; 188(1): 15-22.
8. Keevil CW, Hough JS, Cole ja. „Prototrophic growth of Citrobacter freundii and the biochemical basis for its apparent growth requirements in aerated media.”Journal of General Microbiology. 1997 Jan; 98(1): 273-6.
9. Marco Sánchez F, Turabian Fernández JL, Durán Pérez-Navarro A. „Fatal Citrobacter Freundii Bronchopneumonia Acquired in the Community in an Uncompromised Patient.”Hiszpański Dziennik Kliniczny. 1985 kwi; 176( 6): 320
10. Julia L. Badger, Monique F. Stins, and Kwang Sik Kim. „Citrobacter freundii Invades and Replicates in Human Brain Microscular Endothelial Cells.” Hinyokika kiyo. Akt Urologica Japonica. 1985 Lipiec; 31( 7): 1159-70.
11. Drelichman V, Band JD. „Bacteremias due to Citrobacter diversus and Citrobacter freundii. Incidence, risk factors, and clinical outcome.”Archiwum chorób wewnętrznych. 1985 Oct; 145( 10): 1808-10.
12. Srinivasan V, nam HM, Sawant AA, Headrick SI, Nguyen LT, Oliver Sp. „Distribution of Tetracycline and Streptomycin Resistance Genes and Class 1 Integrons in Enterobacteriaceae Isolated from Dairy and Nondairy Farm Soil.”Ekologia Mikrobiologiczna. 2007 Sie 15
13. Sánchez-Céspedes J, Vila J. „Partial characterisation of the acrAB locus in two Citrobacter freundii clinical isolates.”International Journal of Antimicrobial Agents. 2007 Sep; 30(3): 259-63.
14. Ciğerci IH, Korcan SE, Konuk M, Oztürk S. „Comparison of Alad activity of Citrobacter and Pseudomonas strains and their usage as biomarker for PB contamination.”Monitoring i ocena środowiska. 2007 maj 22
15. Chocat P, Esaki N, Tanizawa K, Nakamura K, Tanaka H ,Soda K. ” Purification and characterization of selenocysteine beta-lyase from Citrobacter freundii.”Journal of Bacteriology. 1985 Aug; 163 (2): 669-76.
16. Hillel S Levinson, Inga Mahler. „Aktywność fosfatazy i oporność na ołów w Citrobacter freundii i Staphylococcus aureus.”FEMS Microbiology Letters. 1998 Apr; 161(1): 135-138.
17. Murakami K, Yoshida T. ” Covalent binding of moxalactam to cefalosporinase of Citrobacter freundii.”Środki przeciwdrobnoustrojowe i chemioterapia. 1985 May; 27 (5): 727-32.
18. Dervisoglu, E., Yegenaga, I., Yumuk ,Z. ” Citrobacter Freundii peritonitis and tunnel infection in a patient on continuous ambulatory peritoneal dialize.”Journal of Medical Microbiology, 2008. Tom 57. S. 125-127.
19. Kim PW, Harris AD, Roghmann MC, Morris JG Jr, Strinivasan a, Perencevich EN. „Epidemiological risk factors for isolation of Ceftriaxone-resistant versus-sensible citrobacter freundii in hospitalized patients” Antimicrob Agents Chemother, 2003 Sep;47(9): p. 2882-2887.
20. Nada T, Baba H, Kawamura K, Ohkura T, Torii K, Ohta M. „a small outbreak of third generation cephem-resistant Citrobacter freundii infection on a surgical ward” JPN J Infect Dis. 2004 Aug;57 (4): s. 181-182.
21. Chuang, Y., Tseng, S., Teng, L., Ho, Y., and Hsueh, P. „Emergence of Cefotaxime resistance in Citrobactor freundii causing necrotizing fasciitis and osteomyelitis” Journal of Infection, 2006. Tom 53. P. e161-e163.
22.Badger, J., Stins, M., and Sik Kim, K. „Citrobacter freundii Invades and Replicates in Human Brain Microvascular Endothelial Cells” Infection and Immunity, 1999. Volume67 P. 4208-4215.
pod redakcją Sumairy Akbarzady, studentki Rachel Larsen pod redakcją Grega Vargasa i Darrena Zhena ,studentki Uniwersytetu m Glogowskiat Loyola pod redakcją Gergany Grigorova i Michała Olszewskiego, studentki Uniwersytetu m Glogowskiat Loyola pod redakcją Alexa kuli i Joanny Aiken, studentki Uniwersytetu m Glogowskiat Loyola